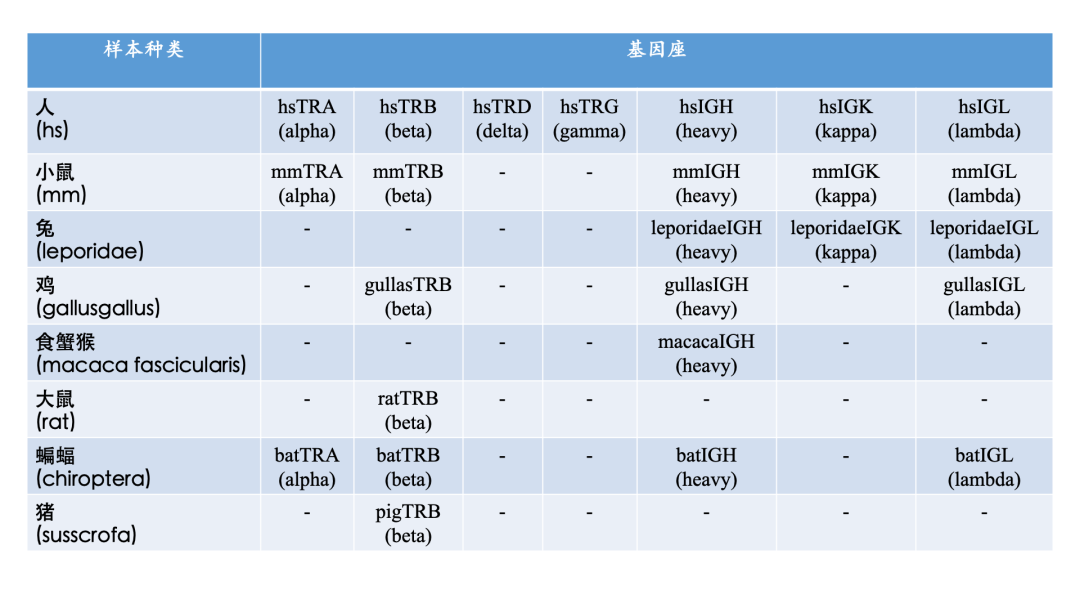
дЇМдї£жµЛеЇПжКАжЬѓеЬ®ињЗеОїдЇМеНБеєідЄ≠еПСе±ХињЕйАЯеєґйАРжЄРжЩЃеПКпЉМTCRж£АжµЛзЪДзБµжХПеЇ¶еТМTCRеПСзО∞еЈ•еЕЈжАІиГљеЊЧеИ∞жЮБе§ІжПРеНЗгАВдЇМдї£жµЛеЇПдЄїи¶БеМЕжЛђDNA/RNAжПРеПЦгАБжЦЗеЇУжЮДеїЇгАБдЄКжЬЇжµЛеЇПгАБжХ∞жНЃиЊУеЗЇињЩеЗ†дЄ™жµБз®ЛгАВеЕґдЄ≠пЉМDNA/RNAжЦЗеЇУжЮДеїЇжШѓдЇМдї£жµЛеЇПжКАжЬѓзЪДйЗНи¶БзОѓиКВгАВ
TзїЖиГЮеПЧдљУпЉИT cell receptorпЉМTCRпЉЙжШѓTзїЖиГЮи°®йЭҐиіЯиі£зЙєеЉВжАІиѓЖеИЂдЄОMHCпЉИдЄїи¶БзїДзїЗзЫЄеЃєжАІе§НеРИдљУпЉЙзїУеРИзЪДжКЧеОЯиВљзЪДиЫЛзЩљгАВељУTCRдЄОжКЧеОЯиВљеТМMHCзїУеРИжЧґпЉМTжЈЛеЈізїЖиГЮйАЪињЗдњ°еϣ蚐僊襀жњАжіїпЉМеРѓеК®еЕНзЦЂеЇФз≠ФгАВдЇЇз±їеЯЇеЫ†зїДдЄ≠жЬЙ4дЄ™TCRеЯЇеЫ†пЉЪдЄ§дЄ™зЉЦз†БиљїйУЊTCRпЉЪTRAеЯЇеЫ†зЉЦз†БTCRќ±пЉМTRGеЯЇеЫ†зЉЦз†БTCRќ≥пЉЫдЄ§дЄ™зЉЦз†БйЗНйУЊTCRпЉЪTRBеЯЇеЫ†зЉЦз†БTCRќ≤пЉМTRDеЯЇеЫ†зЉЦз†БTCRќіпЉЫйЗНйУЊTCRеТМиљїйУЊTCR嚥жИРеЉВжЇРдЇМиБЪдљУпЉМзїДжИРеЃМжХізЪДTCRгАВеЬ®дЇЇз±їдЄ≠е≠ШеЬ®дЄ§зІНTCRпЉЪTCRќ±ќ≤еТМTCRќ≥ќіпЉМеЕґдЄ≠95пЉЕзЪДTзїЖиГЮи°®иЊЊTCRќ±ќ≤гАВжИРзЖЯзЪДйЗНйУЊTCRеЯЇеЫ†зФ±еПѓеПШеМЇпЉИVпЉЙгАБе§ЪеПШеМЇпЉИDпЉЙгАБињЮжО•еМЇпЉИJпЉЙеТМжБТеЃЪеМЇпЉИCпЉЙеЫЫйГ®еИЖеЯЇеЫ†зЙЗжЃµзїДжИРпЉИVDJCпЉЙпЉМиљїйУЊTCRеИЩзЉЇе∞СDеМЇпЉИVJCпЉЙгАВйЗНйУЊеТМиљїйУЊTCRйГљеЕЈжЬЙ3дЄ™дЇТи°•еЖ≥еЃЪеМЇпЉИCDRпЉЙпЉМCDRеЬ®жКЧеОЯиѓЖеИЂдЄ≠иµЈдЄїи¶БдљЬзФ®пЉЪCDR1еТМCDR2зЫЄеѓєдњЭеЃИпЉМиіЯиі£иѓЖеИЂMHCпЉЫCDR3жШѓдЄОжКЧеОЯзЫіжО•жО•иІ¶зЪДTCRеМЇеЯЯпЉМCDR3зФ±дЄАйГ®еИЖVгАБеЕ®йГ®DеТМJдї•еПКV-DгАБD-JдєЛйЧіињЮжО•еМЇзЉЦз†БпЉМеЫ†ж≠§CDR3еПШеМЦз®ЛеЇ¶жЬАйЂШгАВзФ±дЇОVпЉИ65~100зІНпЉЙгАБDпЉИ2зІНпЉЙгАБJпЉИ13зІНпЉЙеЯЇеЫ†зЙЗжЃµжЬђиЇЂеЕЈжЬЙе§Ъж†ЈжАІпЉМж≠§е§ЦпЉМеЬ®йЗНжОТзЪДињЗз®ЛдЄ≠пЉМVDеПКD-JзЪДињЮжО•еМЇзїПеЄЄжЬЙйЭЮж®°жЭњзЪДж†ЄиЛЈйЕЄйЪПжЬЇжПТеЕ•жИЦеИ†йЩ§пЉМињЫдЄАж≠•еҐЮеК†дЇЖCDR3еМЇзЪДе§Ъж†ЈжАІпЉМзРЖиЃЇдЄКдЉЪ嚥жИР2√Ч1019зІНTCRќ±ќ≤гАВ
зФ±дЇОеЬ®дЄАдЄ™дЄ™дљУеЖЕеЗ†дєОдЄНдЉЪеЗЇзО∞дЄ§дЄ™еЃМеЕ®зЫЄеРМзЪДVпЉИDпЉЙJдљУзїЖиГЮйЗНжОТпЉМжЙАдї•TCRеЇПеИЧеПѓдљЬдЄЇTзїЖиГЮеЕЛйЪЖзЪДзЛђзЙєиѓЖеИЂж†ЗењЧгАВTCRињЩзІНзЙєжАІеПѓзФ®дЇОиѓДдЉ∞пЉИ1пЉЙжКЧеОЯй©±еК®зЪДTзїЖиГЮеЕЛйЪЖжЙ©еҐЮеТМеПНеЇФпЉИеЫЊ1aеТМdпЉЙпЉИ2пЉЙзЇµеРСзЫСжµЛTзїЖиГЮеПНеЇФзЪДеЕЛйЪЖеК®жАБеТМеЉВиі®жАІпЉИеЫЊ1bеТМcпЉЙпЉИ3пЉЙеЬ®еНХдЄ™зїЖиГЮдЄ≠иБФеРИиѓДдїЈTCRеТМзїЖиГЮи°®еЮЛиГље§ЯжПРдЊЫTзїЖиГЮеИЖеМЦйАЪиЈѓеТМTзїЖиГЮйАЙжЛ©зЪДзЫЄеЕ≥дњ°жБѓпЉИеЫЊ1bпЉЙгАВињЩдЇЫдњ°жБѓдЄНдїЕжЬЙеК©дЇОзРЖиІ£еЕНзЦЂзЫЄеЕ≥зЦЊзЧЕзЪДзЧЕеЫ†е≠¶пЉМдєЯеПѓзФ®дЇОиЃЊиЃ°ж≤їзЦЧйЭґзВєгАВ
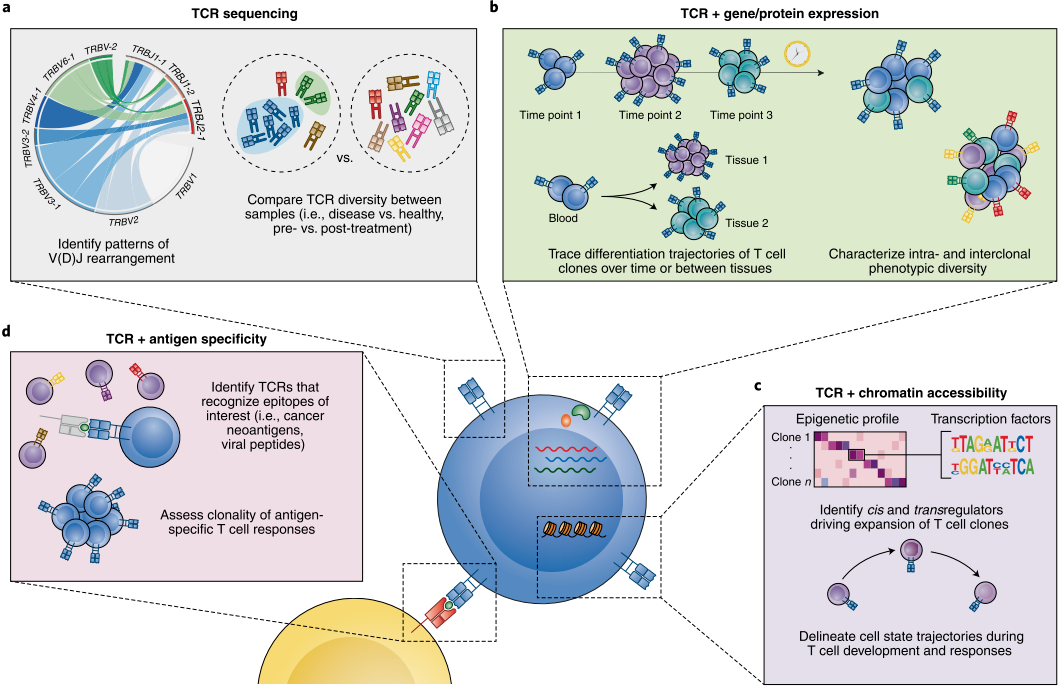
вЦ≤ еЫЊ1. еИ©зФ®TCRе§ЪзїДе≠¶жµЛеЇПжЦєж≥ХеИїзФїTзїЖиГЮеК®жАБ
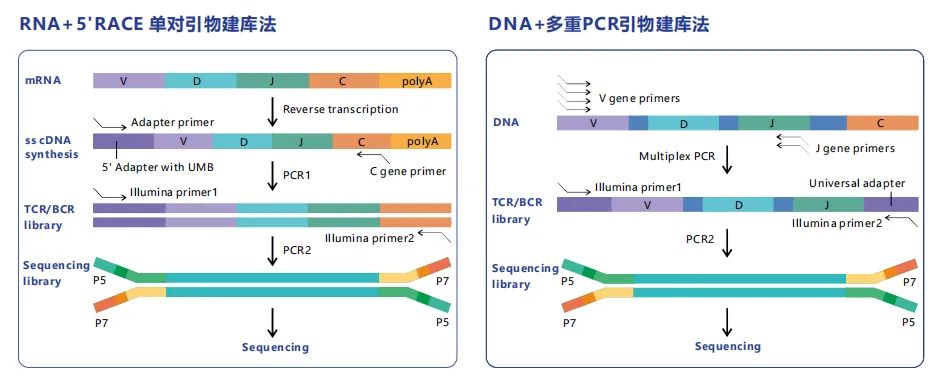
еИ©зФ®йЂШйАЪйЗПжµЛеЇПжКАжЬѓињЫи°МTCRеЇУеИЖжЮРпЉМеПѓдї•еЃЮзО∞еЬ®дЄАжђ°еЃЮй™МдЄ≠еРМжЧґеИЖжЮРжХ∞дї•зЩЊдЄЗиЃ°зЪДTзїЖиГЮпЉИеЫЊ1aпЉЙгАВињЩдЇЫжЦєж≥ХйАЪеЄЄйЗЗзФ®дї•дЄЛдЄ§зІНжЙ©еҐЮз≠ЦзХ•дЄ≠зЪДдЄАзІНпЉЪпЉИ1пЉЙе§ЪйЗНPCRпЉИ2пЉЙ5вАЩдЇТи°•зЂѓењЂйАЯжЙ©еҐЮпЉИ5вАЩRACEпЉЙгАВ
е§ЪйЗНPCR
зФ±дЇОTCR VеЯЇеЫ†еЈ®е§ІзЪДе§Ъж†ЈжАІпЉМдЄАеѓєеЉХзЙ©дЄНиґ≥дї•жНХиОЈжЙАжЬЙзЪДTCRиљђељХжЬђпЉМжЙАдї•еЗЇзО∞дЇЖе§ЪйЗНPCRеПНеЇФгАВе§ЪйЗНPCRжЦєж≥ХдљњзФ®дЄАзїДдЄОеЈ≤зЯ•VеЯЇеЫ†дЇТи°•зЪДж≠£еРСеЉХзЙ©еТМдЄАзїДдЄОJжИЦCеМЇеЯЯдЇТи°•зЪДеПНеРСеЉХзЙ©гАВеЬ®ињЩзІНеЉХзЙ©жЙ©еҐЮз≠ЦзХ•дЄ≠пЉМй¶ЦеЕИдїОTзїЖиГЮдЄ≠еИЖз¶їgDNAжИЦRNAпЉМзДґеРОйЗЗзФ®дЄКињ∞еЉХзЙ©еѓєињЫи°Ме§ЪиљЃPCRжЙ©еҐЮпЉМињЩдЇЫеЉХзЙ©еРМжЧґеРЂжЬЙзФ®дЇОеРОзї≠йЂШйАЪйЗПжµЛеЇПзЪДйАЪзФ®еЇПеИЧгАВRobinsз≠ЙйЗЗзФ®ињЩзІНжЦєж≥ХињЫи°МжЈ±еЇ¶CDR3ќ≤жµЛеЇПеПСзО∞пЉМTCRеЇУеСИзО∞еЗЇзЙєеЃЪV-JйЗНжОТеБПе•љгАВеРМжЧґеПСзО∞пЉМдЄНеРМдЄ™дљУдєЛйЧіеИЭеІЛCDR3ќ≤зЪДйЗНеРИз®ЛеЇ¶ињЬйЂШдЇОеє≥еЭЗж∞іеє≥гАВињЩдЇЫзїУжЮЬи°®жШОTCRеЇУеєґйЭЮйЪПжЬЇдЇІзФЯзЪДпЉМиАМе≠ШеЬ®еѓєжЯРзІНйЗНжОТзЪДеБПе•љпЉМињЩеПѓиГљжШѓзФ±дЇОеЬ®иГЄиЕЇеПСиВ≤ињЗз®ЛдЄ≠зїПеОЖдЇЖеЕ±еРМжКЧеОЯзЪДзЉШжХЕгАВ
ж≠§зІНдЊЭиµЦдЇОе§ЪйЗНPCRжЙ©еҐЮзЪДжЦєж≥ХдЉЪеЉХеЕ•жµЛеЇПеБПе•љеТМйФЩиѓѓпЉМдїОиАМжЧ†ж≥ХиОЈеЊЧTCRе§Ъж†ЈжАІзЪДзЬЯеЃЮжГЕеЖµгАВзЫЃеЙНжЬЙеЗ†зІНз≠ЦзХ•зФ®дЇОеЗПе∞ПињЩзІНеБПе•љпЉМдЊЛе¶ВпЉМзФ±дЇОcDNAиљђељХжЬђеЈ≤зїПжШѓзїПињЗеЙ™еИЗзЪДпЉМжЙАдї•дљњзФ®mRNAеПѓдї•йЗЗзФ®жЫіе∞СзЪДеПНеРСеЉХзЙ©дї•йЭґеРСCеМЇеЯЯпЉМдїОиАМйЩНдљОдЇЖжЭ•иЗ™е§ЪйЗНJеЉХзЙ©PCRжЙ©еҐЮзЪДеБПе•љжАІгАВзЫЄеПНпЉМйЗЗзФ®gDNAжµЛеЇПиГље§ЯйБњеЕНеПНиљђељХпЉМдїОиАМйЩНдљОеЬ®cDNAеРИжИРињЗз®ЛдЄ≠еЉХеЕ•йФЩиѓѓзЪДеПѓиГљгАВж≠§е§ЦпЉМйЗЗзФ®еРИжИРTCRеИЖе≠РйЭґеРСе§ЪйЗНеЉХзЙ©еПѓдї•еЬ®е§ЪйЗНPCRеЙНеРОеЃЪйЗПж®°жЭњпЉМдїОиАМдЉШеМЦеЉХзЙ©жµУеЇ¶еєґж†°ж≠£жЙ©еҐЮеБПе•љгАВ
5вАЩ RACE
зђђдЇМзІНTCRжµЛеЇПжЦєж≥ХжШѓеЯЇдЇО5вАЩRACEпЉМињЩзІНжЦєж≥ХйЬАи¶БдљњзФ®еЕЈжЬЙжЬЂзЂѓиљђзІїйЕґжіїжАІзЪДйАЖиљђељХйЕґеПНиљђељХRNAпЉМдїОиАМеЬ®cDNAзЪД3вАЩзЂѓеК†еЕ•йЭЮж®°жЭњCеѓ°ж†ЄиЛЈйЕЄпЉМзФ®дЇОдЄЛжЄЄзЪДcDNAжЙ©еҐЮпЉМйЭЮж®°жЭњCеѓ°ж†ЄиЛЈйЕЄеПЂеБЪж®°жЭњиљђжНҐеѓ°ж†ЄиЛЈйЕЄпЉИTemplate switch oligoпЉМTSOпЉЙгАВеЬ®зђђдЄАйУЊеРИжИРињЗз®ЛдЄ≠пЉМељУеИ∞иЊЊ RNA ж®°жЭњзЪД 5вАЩжЬЂзЂѓжЧґпЉМйАЖиљђељХйЕґзЪДжЬЂзЂѓиљђзІїйЕґжіїжАІдЉЪеЬ®жЦ∞еРИжИРзЪД cDNA йУЊзЪД3вАЩжЬЂзЂѓжЈїеК†дЄАдЇЫйҐЭе§ЦзЪДж†ЄиЛЈйЕЄгАВињЩдЇЫ祱еЯЇеЕЕељУж®°жЭњиљђжНҐ (TS) еѓ°ж†ЄиЛЈйЕЄйФЪеЃЪдљНзВєгАВеЬ® TS еѓ°ж†ЄиЛЈйЕЄеТМйЩДеК†зЪДиД±ж∞ІиГЮиЛЈзЙЗжЃµдєЛйЧіињЫи°М祱еЯЇйЕНеѓєеРОпЉМйАЖиљђељХйЕґвАЬиљђжНҐвАЭж®°жЭњйУЊпЉМдїОзїЖиГЮ RNA еИ∞ TS еѓ°ж†ЄиЛЈйЕЄпЉМеєґзїІзї≠е§НеИґеИ∞ TS еѓ°ж†ЄиЛЈйЕЄзЪД5вАЩзЂѓгАВињЩзІНжЦєж≥ХеПѓдї•еЊЧеИ∞еМЕеРЂеЃМжХі5вАЩзЂѓзЪДcDNAпЉМињЩжДПеС≥зЭАеПѓдї•дњЭзХЩеЃМжХізЪДVеЯЇеЫ†еЇПеИЧгАВ5вАЩRACEж≥ХйЬАи¶БиЊГе∞СзЪДPCRжЙ©еҐЮпЉМдЄОе§ЪйЗНPCRж≥ХзЫЄжѓФеПѓдї•йЩНдљОжЙ©еҐЮеБПе•љпЉМзДґиАМињЩдЇЫжЦєж≥ХдїНзДґдЉЪе≠ШеЬ®жЭ•иЗ™PCRгАБж®°жЭњиљђжНҐдї•еПКжµЛеЇПзЪДйФЩиѓѓгАВ
TCRжЙ©еҐЮжЦєж≥ХзЪДйФЩиѓѓзЯЂж≠£з≠ЦзХ•
зФ±дЇОдЄКињ∞TCRжµЛеЇПз≠ЦзХ•еПѓиГље≠ШеЬ®зЪДеБПе•љеТМйФЩиѓѓпЉМеЉХеЕ•зЛђзЙєеИЖе≠Рж†Зз≠ЊпЉИUMIsпЉЙеПѓдї•жЬЙжХИзЯЂж≠£ињЩдЇЫйФЩиѓѓгАВUMIsжШѓдЄАз≥їеИЧйЪПжЬЇDNAеЇПеИЧпЉМеЬ®cDNAеРИжИРињЗз®ЛдЄ≠襀еК†еЕ•cDNAдЄ≠еєґж†ЗиЃ∞еФѓдЄАдЄАжЭ°cDNAеИЖе≠РгАВињЩзІНз≠ЦзХ•жЬЙдЄ§дЄ™дЉШеКњпЉЪпЉИ1пЉЙйАЪињЗиЃ°жХ∞жѓПзІНUMIзЪДжХ∞зЫЃеПѓдї•еЊЧеИ∞ж†ЈжЬђдЄ≠TCRеЇПеИЧзЪДеОЯеІЛеИЖеЄГпЉМињЩжЬЙеК©дЇОжЫіз≤Њз°ЃзЪДеЃЪйЗПTCRеЕЛйЪЖйҐСзОЗпЉЫпЉИ2пЉЙйАЪињЗе∞ЖеЕЈжЬЙзЫЄеРМUMIзЪДreadsиБЪйЫЖеЬ®дЄАиµЈеПѓдї•жЬЙжХИзЯЂж≠£PCRеТМжµЛеЇПйФЩиѓѓпЉМдїОиАМеПѓдї•жО®жЦ≠еЗЇзЬЯеЃЮзЪДTCRеЇПеИЧгАВзФ±дЇОTCRsеЇПеИЧдєЛйЧізЪДеЈЃеЉВдїЕжЬЙеЗ†дЄ™ж†ЄиЛЈйЕЄпЉМињЩзІНзЯЂж≠£ж≠•й™§еПѓдї•еМЇеИЖзЬЯеЃЮзЪДTCRеПШеЉВеТМйФЩиѓѓеѓЉиЗізЪДеБЗжАІе§Ъж†ЈжАІпЉМжЙАдї•еПѓдї•жЫіеЗЖз°ЃеЬ∞иѓДдЉ∞TCRеЇУе§Ъж†ЈжАІгАВзДґиАМпЉМиОЈеЊЧеЗЖз°ЃеЇ¶зЪДдї£дїЈжШѓиЊГдљОзЪДзБµжХПеЇ¶пЉМеЫ†дЄЇдљОйҐСеЕЛйЪЖеПѓиГљзФ±дЇОжѓПдЄ™UMIи¶ЖзЫЦзЪДreadдЄНиґ≥иАМ襀ињЗжї§жОЙгАВзЫЃеЙНеЈ≤зїПеЉАеПСеЗЇеѓєжѓПдЄ™ж†ЄиЛЈйЕЄињЫи°МйФЩиѓѓзЯЂж≠£зЪДзЃЧж≥ХпЉМињЩеПѓдї•жЫіз≤Њз°ЃеЬ∞жµЛйЗПTCRеЇУдЄ≠зЪДеЕЛйЪЖеЮЛйҐСзОЗгАВ
TCRйЭґеРСжµЛеЇПжЦєж≥ХзЪДжѓФиЊГ
зФ±дЇОTCRеЇУжµЛеЇПжЦєж≥ХдЄНеРМпЉМжЙАдї•еЬ®еИЖжЮРжµЛеЇПзїУжЮЬжЧґеЇФиАГиЩСдї•дЄЛеЗ†дЄ™еЫ†зі†пЉЪй¶ЦеЕИпЉМжШѓйЗЗзФ®DNAињШжШѓRNAпЉМињЩеЖ≥еЃЪдЇЖеПѓдї•йАВзФ®еУ™зІНжµЛеЇПжЦєж≥ХгАВдЄКињ∞дЄ§зІНжЦєж≥ХеЭЗеПѓзФ®дЇОRNAпЉМзДґиАМпЉМе¶ВжЮЬRNAиі®йЗПжђ†дљ≥пЉМеИЩgDNAе§ЪйЗНжЙ©еҐЮж≥ХеИЩеПѓиГљжШѓжЫіе•љзЪДйАЙжЛ©гАВеЕґжђ°пЉМTCRжµЛеЇПзЪДзЫЃзЪДдєЯдЉЪељ±еУНжµЛеЇПжЦєж≥ХзЪДйАЙжЛ©гАВдЊЛе¶ВпЉМRNAжµЛеЇПзїУжЮЬжЧ†ж≥ХзЫіжО•дЄОзїЖиГЮжХ∞зЫЃзЫЄеЕ≥иБФгАВжЙАдї•пЉМе¶ВжЮЬз†Фз©ґзЫЃж†ЗжШѓеЃЪйЗПжЯРдЄ™TзїЖиГЮзЊ§зЪДеЕЛйЪЖжЙ©еҐЮпЉМеИЩgDNAж≥ХжШѓжЫіе•љзЪДйАЙжЛ©пЉМеЫ†дЄЇжѓПдЄ™зїЖиГЮеП™жЬЙдЄАдЄ™TCRеЯЇеЫ†зїДжЛЈиіЭгАВз±їдЉЉеЬ∞пЉМзФ±дЇОе§ЪйЗНPCRдљњзФ®йЭґеРСVеЯЇеЫ†дЄНеРМеМЇеЯЯзЪДе§ЪзїДеЉХзЙ©пЉМжЙАдї•еЯЇжЬђжЧ†ж≥ХиОЈеПЦиЈ®иґКжХідЄ™VпЉИDпЉЙJеМЇеЯЯзЪДеЕ®йХњTCRеЇПеИЧпЉМињЩзІНжЦєж≥Хиґ≥дї•еИЖжЮРеЕЈжЬЙжЬАйЂШеПШеЉВз®ЛеЇ¶зЪДCDR3еМЇпЉМзДґиАМпЉМе¶ВжЮЬжЛЯеЫЮз≠ФзЪДзФЯзЙ©е≠¶йЧЃйҐШйЬАи¶БиѓДдїЈCDR1еТМCDR2з≠ЙеЕґдїЦеМЇеЯЯпЉМеИЩ5вАЩRACEж≥ХжЫідЄЇеРИйАВгАВ
ж≠§е§ЦпЉМдЄ§зІНTCRжµЛеЇПжЦєж≥ХжѓПдЄАж≠•зЪДжКАжЬѓзЙєзВєдєЯдЉЪеѓєTCRеЇУзїУжЮЬеТМеРОзї≠иІ£йЗКдЇІзФЯеЈ®е§Іељ±еУНгАВеЬ®ињСжЬЯдЄАй°єеѓєе§ЪйЗНPCRеТМ5вАЩRACEињЫи°МTCRжµЛеЇПзЪДжЦєж≥Хе≠¶жѓФиЊГз†Фз©ґдЄ≠пЉМBarennesз≠ЙеЬ®зЫЄеРМзЪДTзїЖиГЮзЊ§дљУж†ЈжЬђдЄ≠пЉМиѓДдїЈдЇЖ9зІНTCRжµЛеЇПжµБз®Лдї•жѓФиЊГеЕґеПѓйЗНзО∞жАІгАБеПѓйЗНе§НжАІдї•еПКзБµжХПеЇ¶гАВдїЦдїђж£АжµЛеИ∞жЦєж≥ХзЙєеЉВжАІзїДеЇУзЙєеЊБеЬ®дЄНеРМйЗНе§НйЧіпЉМе≠ШеЬ®йЂШеЇ¶дЄАиЗіжАІпЉМињЩжДПеС≥зЭАжѓПзІНжЦєж≥ХдЉЪдЇІзФЯзЙєеЃЪзЪДеБПе•љгАВйАЪињЗжѓФиЊГйЗЗзФ®жµБеЉПзїЖиГЮжЬѓеЊЧеИ∞зЪДTRBдљњзФ®зїУжЮЬпЉМдїЦдїђиІВеѓЯеИ∞пЉМзЫЄжѓФдЇОеЯЇдЇОgDNAзЪДе§ЪйЗНPCRжИЦ5вАЩRACEж≥ХпЉМеЯЇдЇОRNAзЪДе§ЪйЗНPCRжЦєж≥ХеСИзО∞еЗЇзЙєеЃЪVеЯЇеЫ†жЫіеБПзЪДжµЛеЇПзїУжЮЬпЉМињЩеПѓиГљеПНжШ†дЇЖRNAиљђељХжЬђдЄ∞еЇ¶еЈЃеЉВеЉХиµЈзЪДжЙ©еҐЮеБПе•љгАВжЧ†иЃЇйЗЗзФ®дљХзІНжЦєж≥ХпЉМиОЈеЊЧзЬЯеЃЮеПНжШ†зФЯзЙ©е≠¶еЕНзЦЂзїДеЇУзЪДжµЛеЇПзїУжЮЬеЊИе§Із®ЛеЇ¶дЄКеПЦеЖ≥дЇОиµЈеІЛж†ЄйЕЄйЗПпЉМе∞§еЕґжШѓеЬ®ж£АжµЛзљХиІБTCRеЇПеИЧжЦєйЭҐгАВе¶ВжЮЬдЄАй°єз†Фз©ґзЪДзЫЃж†ЗжШѓиОЈеПЦжЬАе§ІTCRе§Ъж†ЈжАІжИЦж£АжµЛзљХиІБеЕЛйЪЖеЮЛпЉМеИЩеЇФдЉШеЕИиАГиЩСйАЙжЛ©йЗЗзФ®йЭЮUMIзЪД5вАЩRACEж≥ХпЉМеЫ†дЄЇињЩзІНжЦєж≥ХжѓФеЯЇдЇОUMIзЪДжЦєж≥ХзБµжХПеЇ¶жЫійЂШпЉМе∞§еЕґжШѓеѓєдЇОTCRќ±йУЊгАВзЫЄеПНпЉМе¶ВжЮЬзЫЃж†ЗжШѓжЮДеїЇTCRеЇУзЪДдї£и°®жАІеЕЛйЪЖзїУжЮДжИЦиАЕжШѓеЬ®жЙ©еҐЮж∞іеє≥еЯЇз°АдЄКиѓЖеИЂжДЯеЕіиґ£зЪДTCRsпЉМеИЩеЄ¶UMIзЪД5вАЩRACEж≥ХдєЯиЃЄжЫіеРИйАВпЉМеЫ†дЄЇињЩзІНжЦєж≥ХжЫіењ†еЃЮеЬ∞дњЭзХЩTCRеЕЛйЪЖеЮЛйҐСзОЗпЉМе∞љзЃ°йЬАи¶БеЗ†дЄ™йЗНе§НжИЦиАЕжЫійЂШзЪДжµЛеЇПжЈ±еЇ¶дї•жНХиОЈдљОйҐСеЕЛйЪЖгАВ
ImmuHub¬ЃжШѓзФ±иЙЊж≤РиТљеЉАеПСзЪДдЄАе•ЧзБµжіїеЇ¶жЮБйЂШзЪД TCR еТМ BCR дЇМдї£жµЛеЇПеє≥еП∞гАВеПѓж†єжНЃдЄНеРМж†ЈжЬђз±їеЮЛеТМз†Фз©ґйЬАж±ВдЄЇеЃҐжИЈжПРдЊЫдЄ™жАІеМЦзЪДжЦЗеЇУжЮДеїЇеТМжµЛеЇПж®°еЉПпЉМеЕґдЄ≠еМЕжЛђ RNA+5вАЩRACEпЉМRNA+е§ЪйЗН PCR еТМ DNA+е§ЪйЗН PCR жЦєж≥ХпЉМеєґйЕНеРИдЄЦзХМйҐЖеЕИзЪДзФЯзЙ©дњ°жБѓе≠¶зЃЧж≥ХжПРдЊЫеПѓиІЖеМЦжХ∞жНЃеИЖжЮРгАВиЙЊж≤РиТљйАЪеЄЄдљњзФ®RNA+5вАЩRACEеТМDNA+е§ЪйЗН PCRжЦЗеЇУжЮДеїЇжЦєж≥ХпЉМжО®иНРдљњзФ®RNA+5вАЩRACEжЦЗеЇУжЮДеїЇжЦєж≥ХпЉИеЫЊ2гАБеЫЊ3пЉЙгАВ

вЦ≤ еЫЊ2. ImmuHub¬ЃжКАжЬѓжЦєж≥ХдЄОдЉШеКњ
вЦ≤ еЫЊ3. ImmuHub¬ЃжКАжЬѓиЈѓзЇњ
еЬ®RNA+5вАЩRACEжЦЗеЇУжЮДеїЇжЦєж≥ХдЄ≠пЉМиЙЊж≤РиТљеЉХеЕ•UMBпЉИUnique Molecular BarcodeпЉМзЛђзЂЛеИЖе≠Ржݰ嚥з†БпЉЙињЫи°МжЦЗеЇУжЮДеїЇгАВжѓПдЄ™ж†ЈжЬђж£АжµЛдЄ≠пЉМеЬ®еОЯеІЛRNAж®°жЭњжЙ©еҐЮеЙНеК†еЕ•зЪДUMBзІНз±їеПѓйЂШиЊЊ1700дЄЗзІНпЉМињЩеПѓеѓєеРОзї≠PCRеПНеЇФжИЦжµЛеЇПињЗз®ЛдЄ≠зЪДйФЩиѓѓињЫи°МжЬЙжХИзЇ†ж≠£пЉМињШеОЯзЬЯеЃЮзЪДжХ∞жНЃпЉМеЃМеЕ®жЭЬзїЭPCRйФЩиѓѓеТМжµЛеЇПйФЩиѓѓпЉМдїОиАМжПРйЂШеЗЖз°ЃзОЗгАВж®°жЛЯжЧ†PCRеМЦжµЛеЇПпЉМињШеОЯPCRдєЛеЙНзЪДзЬЯеЃЮеИЖе≠РжХ∞йЗПгАВ
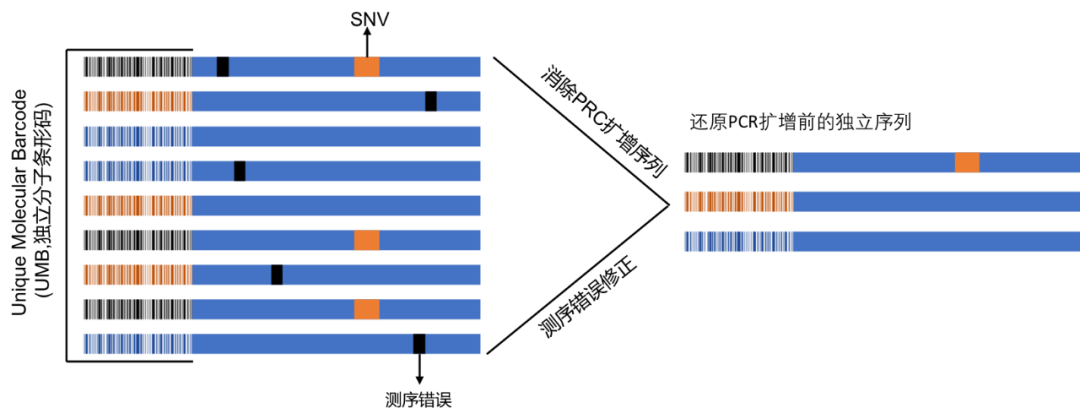
вЦ≤ еЫЊ4. еЯЇдЇОUMBзЪДзЇ†йФЩ
дїАдєИжШѓImmuHub¬ЃжКАжЬѓпЉЯ
зВєеЗїйШЕиѓїеОЯжЦЗпЉМдЇЖиІ£жЫіе§ЪжЬЙеЕ≥ImmuHub¬Ѓ
еПВиАГжЦЗзМЃпЉЪ
[1] Pai JA, Satpathy AT. High-throughput and single-cell T cell receptor sequencing technologies. Nat Methods. 2021;18(8):881-892. doi:10.1038/s41592-021-01201-8
жЭ≠еЈЮиЙЊж≤РиТљзФЯзЙ©зІСжКАжЬЙйЩРеЕђеПЄзФ±зЊОеЫљиКЭеК†еУ•е§Іе≠¶зІСз†ФеЫҐйШЯеЫЮеЫљеИЫеКЮпЉМжШѓдЄАеЃґдЄУж≥®дЇОйАЪињЗиІ£з†БйАВеЇФжАІеЕНзЦЂз≥їзїЯжЭ•жФєеПШзЦЊзЧЕзЪДиѓКжЦ≠еТМж≤їзЦЧпЉМеєґиЗіеКЫдЇОжО®ињЫеЕНзЦЂй©±еК®еМїе≠¶йҐЖеЯЯеПСе±ХзЪДеЫљеЃґйЂШжЦ∞жКАжЬѓдЉБдЄЪгАВиЙЊж≤РиТљзЂЩеЬ®йАВеЇФжАІеЕНзЦЂз≥їзїЯз†Фз©ґзЪДжЬАеЙНж≤њпЉМиЗ™дЄїз†ФеПСзЪДеЕНзЦЂеМїе≠¶еє≥еП∞еПѓжП≠з§ЇеТМзњїиѓСйАВеЇФжАІеЕНзЦЂз≥їзїЯзЪДйБЧдЉ†еѓЖз†БпЉМеєґиГљеЇФзФ®дЇОзЩМзЧЗгАБиЗ™иЇЂеЕНзЦЂжАІзЦЊзЧЕгАБдЉ†жЯУжАІзЦЊзЧЕз≠ЙеЕНзЦЂдїЛеѓЉжАІзЦЊзЧЕзЪДиѓКжЦ≠гАБзЫСжµЛеТМж≤їзЦЧдЄ≠гАВ

