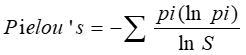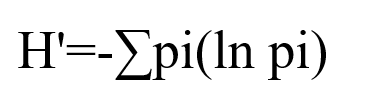ImmuHub® | 艾沐蒽生物免疫组库测序科研服务分析报告
项目信息
| 客户姓名 | XXX |
|---|---|
| 客户单位 | XXXX医院 |
| 合同编号 | / |
| 样本数量 | 2 |
| 物种信息 | 人 |
| 样本类型 | PBMC |
| 方法学 | 5RACE_UMI |
| 测序链 | TRB |
| 报告日期 | 2025-XX-XX |
| 测序平台及测序模式 | NovaSeq PE150 |
一、ImmuHub®技术平台介绍和服务流程
技术平台介绍
ImmuHub®技术平台是利用5'RACE或多重PCR技术,使我们能直接从基因组RNA或DNA来扩增TCR/BCR的全长序列或CDR3区序列,从而进行高通量测序和数据分析。ImmuHub®是艾沐蒽开发的一套灵活度极高的TCR和BCR二代测序平台。
ImmuHub®技术从实验设计到出具数据报告,为您的样本提供一个能获得全长序列或定量的免疫组测序解决方案,这个解决方案使您有能力解读适应性免疫系统的复杂性,帮助您发现适应性免疫系统的广度和深度,从而帮助您在肿瘤的生物标志物,治疗靶点,疗效监测及预后等应用研究中有更好的见解。
技术平台介绍
技术路线
当前使用5RACE_UMI技术方法,5'RACE或多重PCR技术路线图分别如下图所示:
技术路线图
服务流程
对客户送来的PBMC样本提取获得 RNA,对RNA样本进行质检,合成cDNA,使用 ImmuHub® 5′RACE PCR技术法扩增TRB全长基因。回收目的片段,并分别用Qubit、Agilent 2100或者4200和Q-PCR法对文库的浓度、片段的完整性及插入片段大小、文库有效浓度进行检测和精准定量,使用Illumina高通量测序平台NovaSeq®/NextSeq®对TCR文库进行测序,再对下机测序数据进行质量评估,最后使用世界领先的生物信息学分析算法对原始数据(raw data)进行分析和处理。
Methods[1][2][3][4][25][28][29]
RNA samples were analyzed by High-throughput sequencing of TRB using the ImmuHub® TCR profiling system at a deep level (ImmuQuad Biotech, Hangzhou China). Briefly, a 5’ RACE unbiased amplification protocol was used. This protocol uses unique molecular barcodes (UMBs) introduced in the course of cDNA synthesis to control bottlenecks and to eliminate PCR and sequencing errors. Sequencing was performed on an Illumina NovaSeq®/NextSeq® system with PE150/PE300 mode (Illumina). One common adaptor with UMB was added on the 5’ of cDNA during the first-strand cDNA synthesize and one reverse primer corresponding to the constant (C) regions of each of the TRB were designed to facilitate PCR amplification of cDNA sequences in a less biased manner. The UMB attached to each raw sequence reads were applied to correct PCR and sequencing errors correction and PCR duplicates removing. Map V, D, J and C segments with NCBI and then extract CDR3 regions and assemble clonotype for all clones. The resulting nucleotide and amino acid sequences of CDR3 of TRB were determined and those with out-of-frame and stop codon sequences were removed from the identified TRB repertoire. We further defined amounts of each TRB clonotype by adding numbers of TRB clones sharing the same nucleotide sequence of CDR3.
二、生物信息学分析
1.原始数据生物信息学分析
TCR测序只分析覆盖CDR3区域的reads,原始fastq数据需要经过质控处理,测序的原始数据 (raw data)包含低质量、接头污染以及未知碱基N含量过高的reads,去除这部分reads 后,进行后续分析。
原始数据 raw data 为双端测序数据 FASTQ 格式,其中包含测序序列(reads)的序列信息以及其对应的测序质量信息。统计每个样品数据的Q20和Q30,这反映测序数据具有较好的测序质量。
FASTQ 格式示例图
每个 reads 在 FASTQ 文件中表示为 4 行数据:
(1)第一行:reads 名称;以 @ 开头;
(2)第二行:测序得到的 reads 序列;
(3)第三行:'+',无意义;
(4)第四行:reads 每个碱基的质量。
第四行中每个字符对应的 ASCII 值减去 33,即为对应第二行碱基的测序质量值。如果测序错误率用 E 表示,Illumina NovaSeq/NextSeq 的碱基质量值用 Qphred 表示,则有关系:Qphred = -10log10
2.参考受体库比对
过滤后数据与数据库NCBI网站(https://www.ncbi.nlm.nih.gov/)序列比对。
三、标准数据信息分析
1.CDR3克隆鉴定、计数和频率
对比NCBI数据库,分别对每个样本的V、D、J、C片段数据结果进行匹配,使用UMB分析,去除或修正PCR扩增及高通量测序过程中引入的错误序列,同时,合并相同UMB序列,对得到的可信序列计数。获得每条序列中V、D、J、C基因片段ID,然后在具有合格的 CDR3 区域的基础上,鉴定出克隆的CDR3核酸序列以及氨基酸序列,并对得到的每种克隆进行计数和频率的计算。
VDJ全长克隆序列分析详见13.全长克隆序列分析。
文件示例如下表所示:
| Clone.ID | Clone.Count | Clone.Fraction | V.Segment | D.Segment | J.Segment | C.Segment | CDR3.Gene.Sequence | CDR3.AA.Sequence |
|---|---|---|---|---|---|---|---|---|
| 1 | 52061 | 0.100488 | TRBV5-1 | NaN | TRBJ1-1 | TRBC1 | TGCGCCAGCAGCCCAGAACAGCTGGCTAGAAGCACTGAAGCTTTCTTT | CASSPEQLARSTEAFF |
| 2 | 22100 | 0.042657 | TRBV4-3 | TRBD2 | TRBJ2-7 | TRBC2 | TGCGCCAGCAGCCAAGATTATACTAGCGGGAGCCCCTACGAGCAGTACTTC | CASSQDYTSGSPYEQYF |
| 3 | 11212 | 0.021641 | TRBV4-3 | TRBD1 | TRBJ2-3 | TRBC2 | TGCGCCAGCAGCCAAGAGGCGCCAGATACGCAGTATTTT | CASSQEAPDTQYF |
| 4 | 6495 | 0.012537 | TRBV5-1 | TRBD1 | TRBJ1-1 | TRBC1 | TGCGCCAGCAGCCGAGAGCAGACAGGGCGGACTGAAGCTTTCTTT | CASSREQTGRTEAFF |
| 5 | 5486 | 0.010589 | TRBV5-1 | TRBD1 | TRBJ2-7 | TRBC2 | TGCGCCAGCAGCTTGGAACAGGCAGCGCGCTCCTACGAGCAGTACTTC | CASSLEQAARSYEQYF |
| 6 | 5438 | 0.010496 | TRBV5-1 | TRBD2 | TRBJ2-3 | TRBC2 | TGCGCCAGCAGCCTGGCTTTAGCGGGGACTAGCACAGATACGCAGTATTTT | CASSLALAGTSTDTQYF |
表头解释:
(1)Clone ID: 每种克隆编号,从1开始按顺序编号
(2)Clone Count: 每种克隆的总数
(3)Clone Fraction: 表示每种克隆占总TCR克隆种类的频率的百分比
(4)V/D/J/C Segment: V/D/J/C 基因片段ID
(5)CDR3 Gene Sequence: CDR3核酸序列
(6)CDR3 AA Sequence: productive CDR3氨基酸序列
2.克隆性指数和多样性指数
克隆性指数和多样性指数表格,文件示例如下表所示:
| FileName | Clonality | Pielous | Shannon.Index | Invsimpson.Index | Simpson.Index | Hvj.Index | Singleton | DE50 | Convergence |
|---|---|---|---|---|---|---|---|---|---|
| cure4_B | 0.240034 | 0.759966 | 8.630488 | 75.585642 | 0.986770 | 4.867105 | 0.103406 | 0.021974 | 0.425949 |
| cure2_B | 0.070538 | 0.929462 | 10.708691 | 3196.798904 | 0.999687 | 5.474696 | 0.166310 | 0.123589 | 0.202016 |
2.1 Clonality Index[5][6][7]
Clonality克隆性指数在于对TCR/BCR克隆扩增的衡量,Clonality值越大,表示有部分克隆发生了很大频率的扩增。公式中,productive uniques 为样本中克隆种类总数,diversity为shannon多样性指数的值。
2.2 Pielou’s Index[8][9][10]
Pielou’s用来评估克隆分布均匀程度,Pielou’s值越高,表示各克隆分布比较均匀, Pielou’s值越低,表示有一些特异性的克隆发生了扩增。公式中,pi为i总序列的比例,S为克隆类型总数。
2.3 Shannon's Index[11][12][13]
香农用来衡量样本中TCR/BCR克隆的多样性。香农指数越高,表示TCR/BCR的多样性越高。该指数能够描述两方面的信息:1.种类丰富度;2.种类中个体分配上的均匀性(evenness)。公式中,pi为第i个特异性克隆类型的频率。可以看出,香农既考虑了TCR/BCR克隆种类的多少,也考虑了每种克隆的均匀度(频率)。
2.4 Simpson's Index(Gini-Simpson)[14][15]
我们通常不直接用Simpson index(D),它反映的是在同一个样本中随机的抽取2个个体,这两个个体来自同一个类的概率。故D值越大,多样性越低。这与直觉和逻辑不符。
为了解决这个问题,通常会用以下两种形式来表示:
(1)1-D,即Gini-Simpson Diversity index
(2)1/D,即Inverse-Simpson index
Gini-Simpson多样性指数表示在克隆群中随机选取两个克隆序列,两者属于不同种类克隆的概率。Gini-Simpson值越高,代表克隆的多样性越高。公式中,ni为第i个特异性克隆类型的氨基酸序列的总数,N为样本中总的序列数。(使用时有些地方也会将Gini-Simpson简化成Simpson,实际上这里是指Gini-Simpson,可根据公式区分)
2.5 Inverse Simpson index[16][17]
逆辛普森多样性指数是辛普森指数的倒数,逆辛普森指数越大,表示克隆的多样性越高。侧重于反映高频克隆的多样性。
2.6 Hvj Index[18][19]
Hvj多样性指数,用来衡量样本中TCR/BCR克隆V-J基因组合使用的多样性。 pi为第i个v-j基因片段组合使用类型的比例。反映的是对T/B细胞的扩增的衡量,也就是反映了T/B特异性识别抗原产生的应激后的复制增殖能力,值越大,表示克隆性扩增越多。
2.7 Singleton[25]
Singleton,用于计算单克隆细胞的比例。例如在TCR中,有研究表明,单克隆T细胞与初始T细胞的比例成正相关,初始T细胞越多,机体识别陌生疾病能力也越强。
2.8 DE50[20][21]
DE50作为克隆性(克隆程度)的一个指标,衡量克隆性的均一程度,DE50值越高,代表各克隆分布比较均匀,DE50值越低,克隆性越高,代表有一些特异性的克隆发生了扩增。公式的计算方法为:将克隆序列按照频率从高到低排序,从最高开始累加,达到频率总和为50%的这些序列的克隆种类占总克隆种类的比例。
2.9 TCR Convergence[30][31]
TCR Convergence 是指不同的V(D)J基因重排过程中,DNA序列由于密码子简并性,可编码相同TCR 氨基酸序列的现象。学术研究表明, 趋同(Convergence)T细胞反映克隆选择的强度,更有可能与抗原特异性有关,被用于评估对治疗的反应有效性。 公式的计算方法为:统计具有相同CDR3AA 和V基因,但不同CDR3核酸序列的克隆频率总和。
3.CDR3核酸序列长度[22]
统计各样本 CDR3 核酸序列的长度,按照频率选取 top10 以及其他(Other)克隆(以CDR3的氨基酸序表示)的 CDR3 核酸长度分布。
核酸序列长度分布柱状图,如下图所示:
图3. CDR3核酸序列长度
4.气泡图
气泡图显示每个克隆种类的频率大小,气泡数越多、越密集表示该样本中克隆种类数越多,中间气泡为Top克隆,面积越大表示频率越大。
气泡图,如下图所示:
图4. 气泡图
5.V/D/J基因频率分布柱状图
对各样本中鉴定到的V/D/J基因片段进行频率计算,分别绘制 V/D/J 基因表达的2D柱状图。
以V基因频率分布柱状图为例,如下图所示:
图5. V/D/J基因频率分布柱状图
6.V/D/J基因频率分布饼状图[23]
对各样本中鉴定到的V/D/J基因片段进行频率计算,分别绘制 V/D/J 基因表达的2D饼状图。
以V基因频率分布饼状图为例,如下图所示:
图6. V/D/J基因频率分布饼状图
7.V-J基因片段组合circos图[14]
V 和 J 基因片段组合图。每一个色块代表一个基因片段,上段为 V 片段,下段为 J 片段,色块越宽代表这个片段使用频率越高,连线越多,表示这种 V-J 组合频率越高。
V-J基因片段组合circos图,如下图所示:
图7. V-J基因片段组合circos图
8.V-J基因片段组合使用森林图[22]
对各样本中鉴定到的V和J基因片段组合进行频率计算,并绘制3D森林图。3D森林图直观的体现T细胞受体(TCR)的克隆性和多样性。每根竖条代表了每种V基因片段和J基因片段重组频率,竖条越多,表示T细胞的V-J组合种类越多,竖条越长,表示这种组合的频率越高。
V-J基因片段组合使用森林图,如下图所示:
图8. V-J基因片段组合使用森林图
9.V-J基因片段组合使用热力图
对各样本中鉴定到的V和J基因片段组合进行频率计算,并绘制热力图,暖色(红色)表示高频,冷色(蓝色)表示低频。连线表示聚类关系,V/J基因使用频率相似性越高,距离相近,则优先聚在一起。
V-J基因片段组合使用热力图,如下图所示:
图9. V-J基因片段组合使用热力图
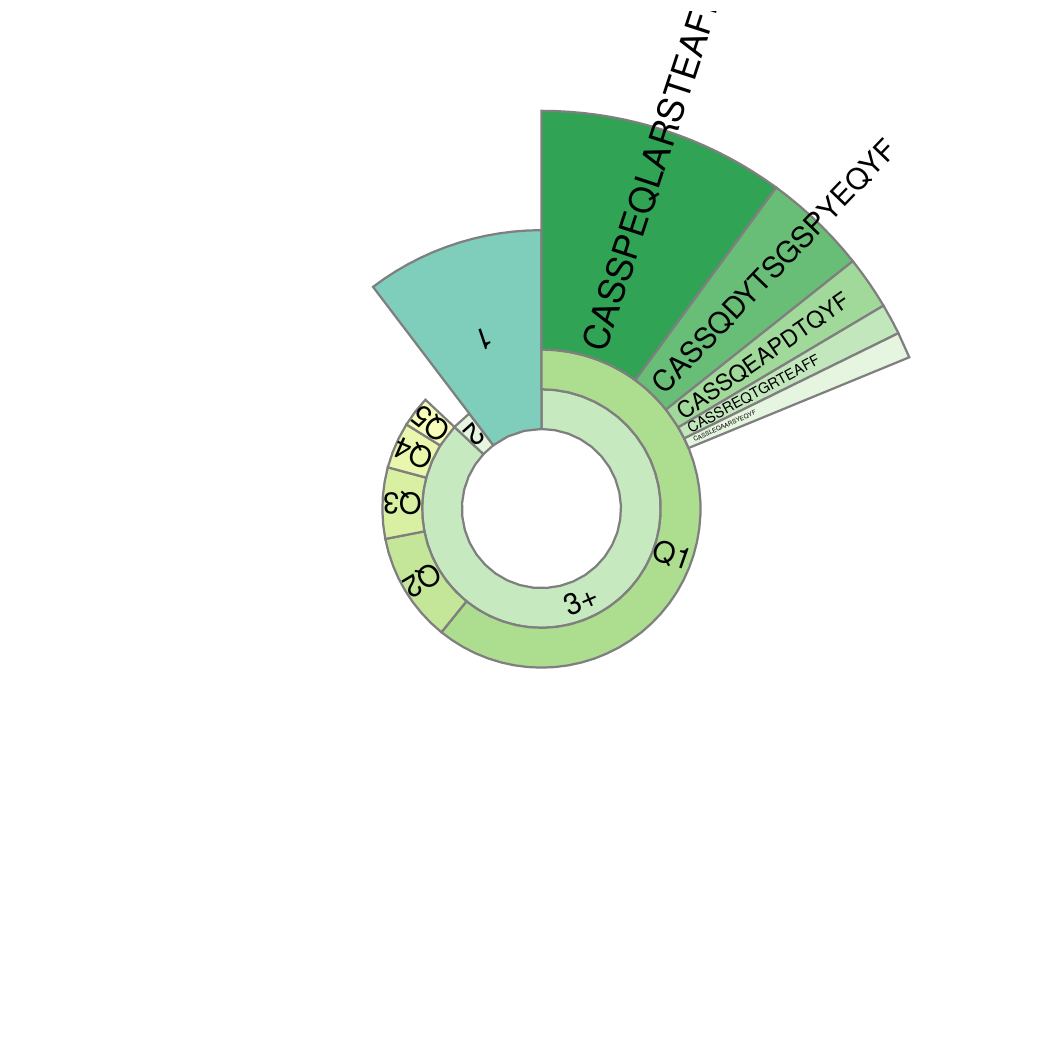
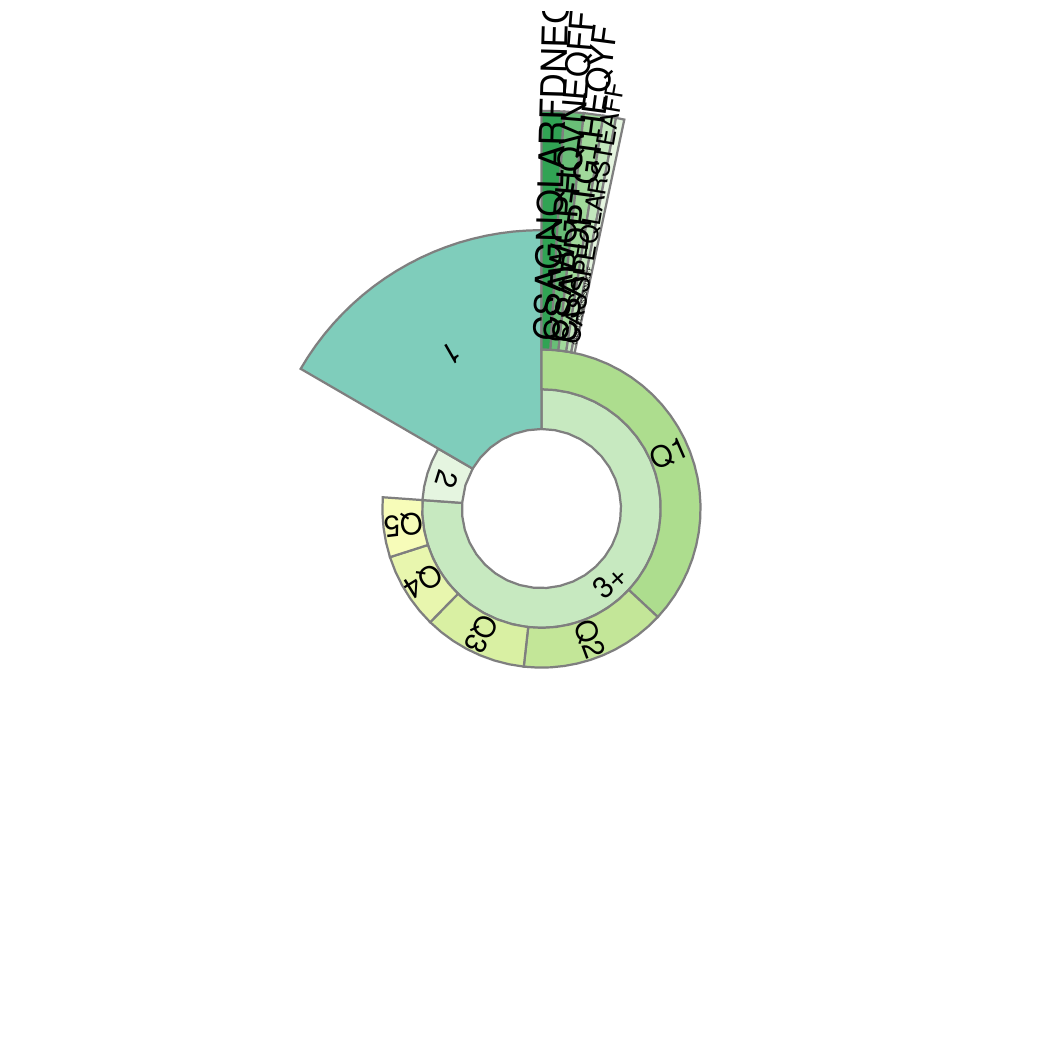
10.克隆频率分类呈现蜗牛图
第一层(最内层)将所有检测到的TCR(100%),分为“1”、“2”和“3+”三类,分别表示检测到了有 1 个、 2 个和3个以上 TCR 分子的 T 细胞克隆序列的比例,内圈弧度越宽,比例越高。“1”为 “单克隆 T 细胞”,它所占的比例是非常重要的,能影响 T 细胞多样性的计算。并且,研究发现[1]“1” 的比例能直观地反映初始 T 细胞(naive T Cell)的比例。初始 T 细胞是免疫系统识别并初始抵御陌生病原体的中坚力量。
第二层将“3+”分为Q1-Q5,分别表示每20%的TCR克隆序列所占的比例(TCR克隆频率由高到低排序),这能直观反映TCR克隆性。
第三层(最外层)是克隆频率排名前 5 种 T 细胞克隆群(Top1~5)。使用CDR3氨基酸序列表示。每个扇面的宽度代表其对应克隆群的频率,扇面越宽说明该TCR克隆性增殖越高。
克隆性蜗牛图,如下图所示:
图10. 克隆频率分类呈现蜗牛图
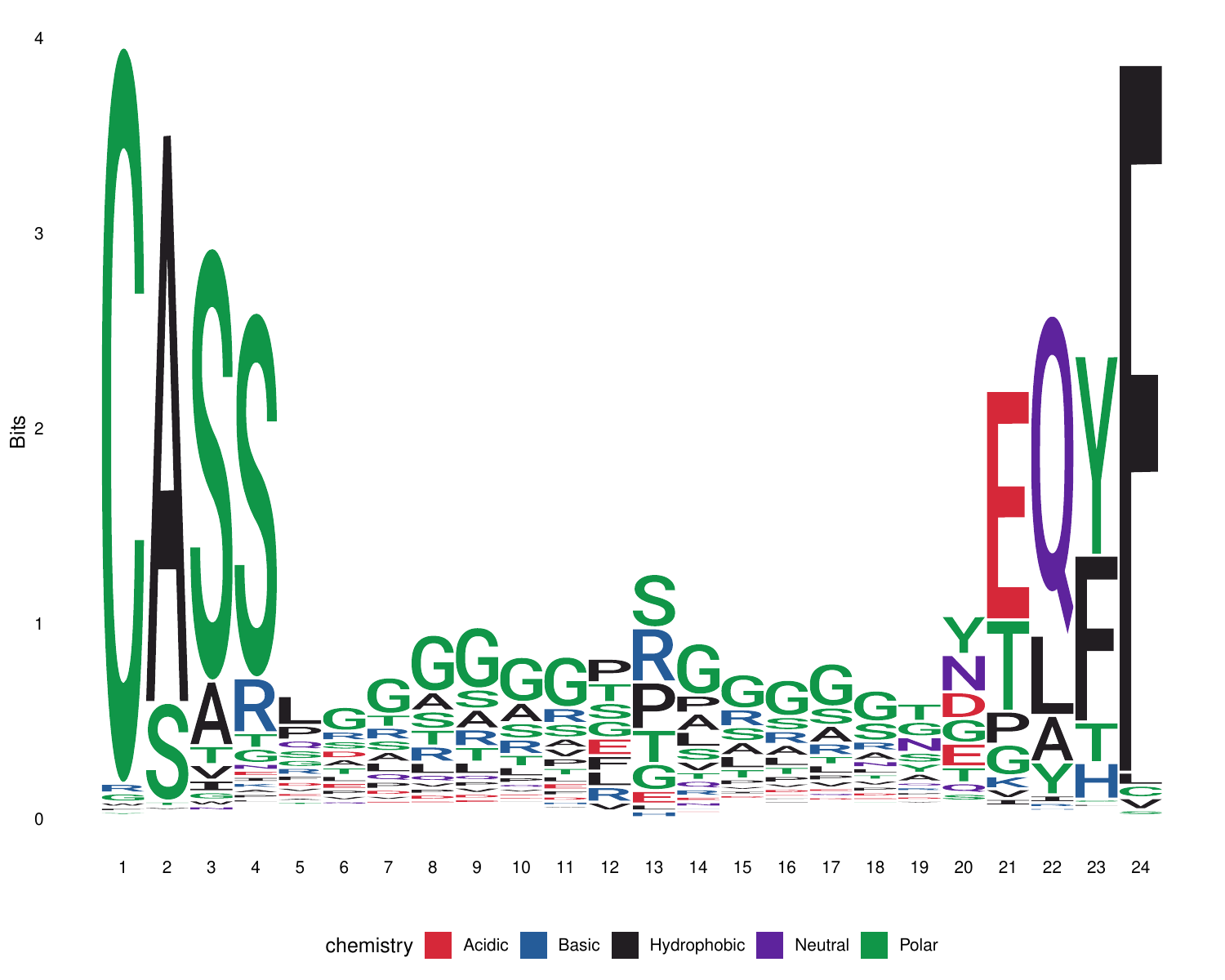
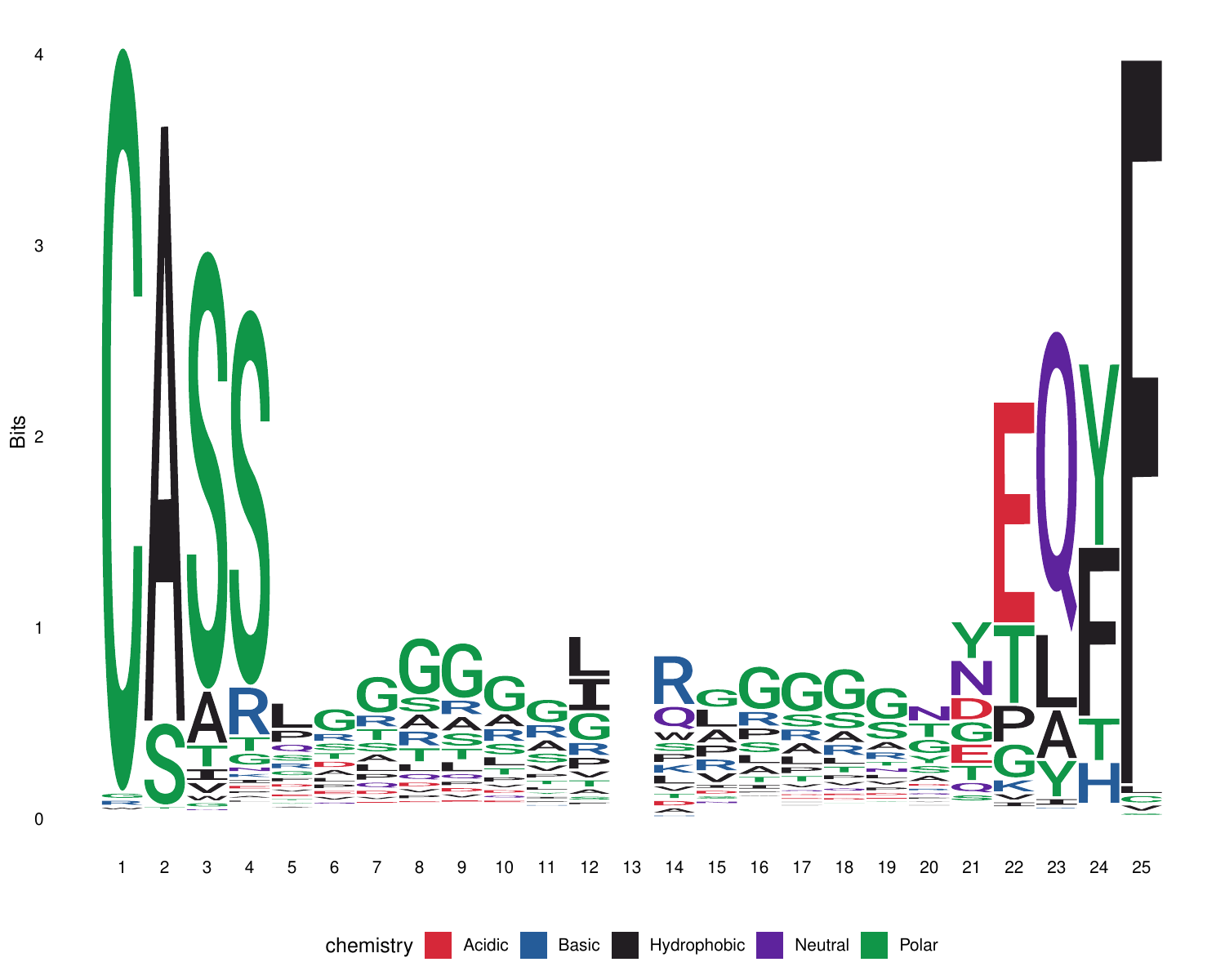
11.CDR3氨基酸保守性分析logo图[26]
CDR3氨基酸的保守性分析有助于理解免疫反应的特异性,以及揭示在不同个体、不同发育阶段或不同免疫状态下,CDR3区域的变异模式,从而理解免疫库的动态变化。
对样本中所有CDR3氨基酸序列进行主要氨基酸来源着色,不同颜色对应不同的氨基酸性质:酸性氨基酸(Acidic)为红色,碱性氨基酸(Basic)为蓝色,疏水氨基酸(Hydrophobic)为黑色,中性氨基酸(Neutral)为紫色,极性氨基酸(Polar)为绿色。横坐标是CDR3氨基酸长度,纵坐标为权重(Bits)。以最长克隆的CDR3长度为基准,其余克隆按照两边对齐的原则聚拢。同一个位置相同的氨基酸越多,字母越大,有多个字母则表示这个位置存在不同的氨基酸。
CDR3氨基酸保守性分析Logo图,如下图所示:
图11. CDR3氨基酸保守性分析logo图
12.TCR-抗原匹配注释
艾沐蒽根据已发表文献的公开数据源建立了ImmuHub®TCR-抗原数据库[32],该数据库包含一些特定疾病的特异性克隆信息。 我们通过和该数据库进行比对,对每个样本的克隆序列进行注释,得到克隆序列和疾病的注释关系。
文件示例如下表所示:
| count | freq | cdr3nt | cdr3aa | vseg | dseg | jseg | cseg | patho | pathology_chinese |
|---|---|---|---|---|---|---|---|---|---|
| 642 | 0.001239 | TGTGCCACCAGCAGAGTGGCAGGAGAGACCCAGTACTTC | CATSRVAGETQYF | TRBV15 | NaN | TRBJ2-5 | TRBC2 | Cytomegalovirus (CMV) | 巨细胞病毒(CMV) |
| 426 | 0.000822 | TGTGCCAGCACCACAGGCGATGAGCAGTTCTTC | CASTTGDEQFF | TRBV25-1 | TRBD1 | TRBJ2-1 | TRBC2 | Cytomegalovirus (CMV) | 巨细胞病毒(CMV) |
| 61 | 0.000118 | TGTGCCAGCAGCTCCGACAGCTACGAGCAGTACTTC | CASSSDSYEQYF | TRBV7-9 | TRBD1 | TRBJ2-7 | TRBC2 | Cytomegalovirus (CMV) | 巨细胞病毒(CMV) |
| 56 | 0.000108 | TGTGCCAGCAGCTTAGGGGGAGCTGAAGCTTTCTTT | CASSLGGAEAFF | TRBV5-6 | TRBD1 | TRBJ1-1 | TRBC1 | Cytomegalovirus (CMV) | 巨细胞病毒(CMV) |
| 41 | 0.000079 | TGTGCCATCAGTGAGGAGACCCCGAACACTGAAGCTTTCTTT | CAISEETPNTEAFF | TRBV10-3 | NaN | TRBJ1-1 | TRBC1 | Cytomegalovirus (CMV) | 巨细胞病毒(CMV) |
| 35 | 0.000068 | TGTGCCAGCAGTCCTGGGGATGAGCAGTTCTTC | CASSPGDEQFF | TRBV25-1 | NaN | TRBJ2-1 | TRBC2 | Cytomegalovirus (CMV) | 巨细胞病毒(CMV) |
| 35 | 0.000068 | TGTGCCAGCAGTCCTGGGGATGAGCAGTTCTTC | CASSPGDEQFF | TRBV25-1 | NaN | TRBJ2-1 | TRBC2 | Influenza | 流感 |
| 34 | 0.000066 | TGCAGTGCTAGACTAGCGGGAGGCCAAGAGACCCAGTACTTC | CSARLAGGQETQYF | TRBV20-1 | TRBD2 | TRBJ2-5 | TRBC2 | M.tuberculosis | 结核分枝杆菌 |
| 32 | 0.000062 | TGTGCCAGCAGTGACTCGAGCACAGATACGCAGTATTTT | CASSDSSTDTQYF | TRBV6-4 | NaN | TRBJ2-3 | TRBC2 | Epstein Barr virus (EBV) | EB病毒(EBV) |
| 32 | 0.000062 | TGTGCCAGCAGTGACTCGAGCACAGATACGCAGTATTTT | CASSDSSTDTQYF | TRBV6-4 | NaN | TRBJ2-3 | TRBC2 | M.tuberculosis | 结核分枝杆菌 |
表头解释:
(1)count: 每种克隆的总数
(2)freq: 每种克隆占总TCR克隆种类的频率的百分比
(3)cdr3nt: CDR3核酸序列
(4)cdr3aa: CDR3氨基酸序列
(5)v/d/j/cseg: VDJC基因片段ID
(6)patho: TCR抗原数据库注释结果
(7)pathology_chinese: TCR抗原数据库注释结果中文
13.全长克隆序列分析
由于本方法是基于5-race扩增技术,上游引物设计在5端,下游引物设计在C端,此流程技术方法可扩增VDJ基因全长,以及C端的引物可以捕获足够的序列以区分亚型。
当使用PE150或PE300双端测序模式时,可以通过测到的基因,与数据库进行匹配,得到V、D、J、C基因片段类型,对介于V、D、J、C片段中间未测到的部分,使用数据库里面的基因进行相应补齐,即得到全长克隆序列(UMB同步分析,如01部分)。
文件示例如下表所示:
| Clone.ID | Clone.Count | Clone.Fraction | V.Segment | D.Segment | J.Segment | C.Segment | V5UTR.Gene.Sequence | L1.Gene.Sequence | L2.Gene.Sequence | FR1.Gene.Sequence | CDR1.Gene.Sequence | FR2.Gene.Sequence | CDR2.Gene.Sequence | FR3.Gene.Sequence | CDR3.Gene.Sequence | FR4.Gene.Sequence | L1.AA.Sequence | L2.AA.Sequence | FR1.AA.Sequence | CDR1.AA.Sequence | FR2.AA.Sequence | CDR2.AA.Sequence | FR3.AA.Sequence | CDR3.AA.Sequence | FR4.AA.Sequence | FullSeq.Gene.Sequence | FullSeq.AA.Sequence |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 52061 | 0.100488 | TRBV5-1 | NaN | TRBJ1-1 | TRBC1 | TATGCCCCTCCCACCAGGAAAAGCAAGGCTGAGAATTTAGCTCTTTCCCAGGAGGACCAAGCCCTGAGCACAGACACAGTGCTGCCTGCCCCTTTGTGCC | ATGGGCTCCAGGCTGCTCTGTTGGGTGCTGCTTTGTCTCCTGGGAGCAG | GCCCAGTA | AAGGCTGGAGTCACTCAAACTCCAAGATATCTGATCAAAACGAGAGGACAGCAAGTGACACTGAGCTGCTCCCCTATC | TCTGGGCATAGGAGT | GTATCCTGGTACCAACAGACCCCAGGACAGGGCCTTCAGTTCCTCTTTGAA | TACTTCAGTGAGACACAG | AGAAACAAAGGAAACTTCCCTGGTCGATTCTCAGGGCGCCAGTTCTCTAACTCTCGCTCTGAGATGAATGTGAGCACCTTGGAGCTGGGGGACTCGGCCCTTTATCTT | TGCGCCAGCAGCCCAGAACAGCTGGCTAGAAGCACTGAAGCTTTCTTT | GGACAAGGCACCAGACTCACAGTTGTAG | MGSRLLCWVLLCLLGA_ | _PS_ | KAGVTQTPRYLIKTRGQQVTLSCSPI | SGHRS | VSWYQQTPGQGLQFLFE | YFSETQ | RNKGNFPGRFSGRQFSNSRSEMNVSTLELGDSALYL | CASSPEQLARSTEAFF | GQGTRLTVV_ | TATGCCCCTCCCACCAGGAAAAGCAAGGCTGAGAATTTAGCTCTTTCCCAGGAGGACCAAGCCCTGAGCACAGACACAGTGCTGCCTGCCCCTTTGTGCCATGGGCTCCAGGCTGCTCTGTTGGGTGCTGCTTTGTCTCCTGGGAGCAGGCCCAGTAAAGGCTGGAGTCACTCAAACTCCAAGATATCTGATCAAAACGAGAGGACAGCAAGTGACACTGAGCTGCTCCCCTATCTCTGGGCATAGGAGTGTATCCTGGTACCAACAGACCCCAGGACAGGGCCTTCAGTTCCTCTTTGAATACTTCAGTGAGACACAGAGAAACAAAGGAAACTTCCCTGGTCGATTCTCAGGGCGCCAGTTCTCTAACTCTCGCTCTGAGATGAATGTGAGCACCTTGGAGCTGGGGGACTCGGCCCTTTATCTTTGCGCCAGCAGCCCAGAACAGCTGGCTAGAAGCACTGAAGCTTTCTTTGGACAAGGCACCAGACTCACAGTTGTAG | MGSRLLCWVLLCLLGAGPVKAGVTQTPRYLIKTRGQQVTLSCSPISGHRSVSWYQQTPGQGLQFLFEYFSETQRNKGNFPGRFSGRQFSNSRSEMNVSTLELGDSALYLCASSPEQLARSTEAFFGQGTRLTVV_ |
| 2 | 22100 | 0.042657 | TRBV4-3 | TRBD2 | TRBJ2-7 | TRBC2 | CATCACAGGAAAAACCACCAACCAAGGCCAAGGAGACCAGAGCCCAGCACCTCACCCAGAGGACCCCAGTCAGAGGCCCCATCTCAGACCCGAGGCTAGC | ATGGGCTGCAGGCTGCTCTGCTGTGCGGTTCTCTGTCTCCTGGGAGCGG | TCCCCATG | GAAACGGGAGTTACGCAGACACCAAGACACCTGGTCATGGGAATGACAAATAAGAAGTCTTTGAAATGTGAACAACAT | CTGGGTCATAACGCT | ATGTATTGGTACAAGCAAAGTGCTAAGAAGCCACTGGAGCTCATGTTTGTC | TACAGTCTTGAAGAACGG | GTTGAAAACAACAGTGTGCCAAGTCGCTTCTCACCTGAATGCCCCAACAGCTCTCACTTATTCCTTCACCTACACACCCTGCAGCCAGAAGACTCGGCCCTGTATCTC | TGCGCCAGCAGCCAAGATTATACTAGCGGGAGCCCCTACGAGCAGTACTTC | GGGCCGGGCACCAGGCTCACGGTCACAG | MGCRLLCCAVLCLLGA_ | _PH_ | ETGVTQTPRHLVMGMTNKKSLKCEQH | LGHNA | MYWYKQSAKKPLELMFV | YSLEER | VENNSVPSRFSPECPNSSHLFLHLHTLQPEDSALYL | CASSQDYTSGSPYEQYF | GPGTRLTVT_ | CATCACAGGAAAAACCACCAACCAAGGCCAAGGAGACCAGAGCCCAGCACCTCACCCAGAGGACCCCAGTCAGAGGCCCCATCTCAGACCCGAGGCTAGCATGGGCTGCAGGCTGCTCTGCTGTGCGGTTCTCTGTCTCCTGGGAGCGGTCCCCATGGAAACGGGAGTTACGCAGACACCAAGACACCTGGTCATGGGAATGACAAATAAGAAGTCTTTGAAATGTGAACAACATCTGGGTCATAACGCTATGTATTGGTACAAGCAAAGTGCTAAGAAGCCACTGGAGCTCATGTTTGTCTACAGTCTTGAAGAACGGGTTGAAAACAACAGTGTGCCAAGTCGCTTCTCACCTGAATGCCCCAACAGCTCTCACTTATTCCTTCACCTACACACCCTGCAGCCAGAAGACTCGGCCCTGTATCTCTGCGCCAGCAGCCAAGATTATACTAGCGGGAGCCCCTACGAGCAGTACTTCGGGCCGGGCACCAGGCTCACGGTCACAG | MGCRLLCCAVLCLLGAVPMETGVTQTPRHLVMGMTNKKSLKCEQHLGHNAMYWYKQSAKKPLELMFVYSLEERVENNSVPSRFSPECPNSSHLFLHLHTLQPEDSALYLCASSQDYTSGSPYEQYFGPGTRLTVT_ |
| 3 | 11212 | 0.021641 | TRBV4-3 | TRBD1 | TRBJ2-3 | TRBC2 | CATCACAGGAAAAACCACCAACCAAGGCCAAGGAGACCAGAGCCCAGCACCTCACCCAGAGGACCCCAGTCAGAGGCCCCATCTCAGACCCGAGGCTAGC | ATGGGCTGCAGGCTGCTCTGCTGTGCGGTTCTCTGTCTCCTGGGAGCGG | TCCCCATG | GAAACGGGAGTTACGCAGACACCAAGACACCTGGTCATGGGAATGACAAATAAGAAGTCTTTGAAATGTGAACAACAT | CTGGGTCATAACGCT | ATGTATTGGTACAAGCAAAGTGCTAAGAAGCCACTGGAGCTCATGTTTGTC | TACAGTCTTGAAGAACGG | GTTGAAAACAACAGTGTGCCAAGTCGCTTCTCACCTGAATGCCCCAACAGCTCTCACTTATTCCTTCACCTACACACCCTGCAGCCAGAAGACTCGGCCCTGTATCTC | TGCGCCAGCAGCCAAGAGGCGCCAGATACGCAGTATTTT | GGCCCAGGCACCCGGCTGACAGTGCTCG | MGCRLLCCAVLCLLGA_ | _PH_ | ETGVTQTPRHLVMGMTNKKSLKCEQH | LGHNA | MYWYKQSAKKPLELMFV | YSLEER | VENNSVPSRFSPECPNSSHLFLHLHTLQPEDSALYL | CASSQEAPDTQYF | GPGTRLTVL_ | CATCACAGGAAAAACCACCAACCAAGGCCAAGGAGACCAGAGCCCAGCACCTCACCCAGAGGACCCCAGTCAGAGGCCCCATCTCAGACCCGAGGCTAGCATGGGCTGCAGGCTGCTCTGCTGTGCGGTTCTCTGTCTCCTGGGAGCGGTCCCCATGGAAACGGGAGTTACGCAGACACCAAGACACCTGGTCATGGGAATGACAAATAAGAAGTCTTTGAAATGTGAACAACATCTGGGTCATAACGCTATGTATTGGTACAAGCAAAGTGCTAAGAAGCCACTGGAGCTCATGTTTGTCTACAGTCTTGAAGAACGGGTTGAAAACAACAGTGTGCCAAGTCGCTTCTCACCTGAATGCCCCAACAGCTCTCACTTATTCCTTCACCTACACACCCTGCAGCCAGAAGACTCGGCCCTGTATCTCTGCGCCAGCAGCCAAGAGGCGCCAGATACGCAGTATTTTGGCCCAGGCACCCGGCTGACAGTGCTCG | MGCRLLCCAVLCLLGAVPMETGVTQTPRHLVMGMTNKKSLKCEQHLGHNAMYWYKQSAKKPLELMFVYSLEERVENNSVPSRFSPECPNSSHLFLHLHTLQPEDSALYLCASSQEAPDTQYFGPGTRLTVL_ |
| 4 | 6495 | 0.012537 | TRBV5-1 | TRBD1 | TRBJ1-1 | TRBC1 | TATGCCCCTCCCACCAGGAAAAGCAAGGCTGAGAATTTAGCTCTTTCCCAGGAGGACCAAGCCCTGAGCACAGACACAGTGCTGCCTGCCCCTTTGTGCC | ATGGGCTCCAGGCTGCTCTGTTGGGTGCTGCTTTGTCTCCTGGGAGCAG | GCCCAGTA | AAGGCTGGAGTCACTCAAACTCCAAGATATCTGATCAAAACGAGAGGACAGCAAGTGACACTGAGCTGCTCCCCTATC | TCTGGGCATAGGAGT | GTATCCTGGTACCAACAGACCCCAGGACAGGGCCTTCAGTTCCTCTTTGAA | TACTTCAGTGAGACACAG | AGAAACAAAGGAAACTTCCCTGGTCGATTCTCAGGGCGCCAGTTCTCTAACTCTCGCTCTGAGATGAATGTGAGCACCTTGGAGCTGGGGGACTCGGCCCTTTATCTT | TGCGCCAGCAGCCGAGAGCAGACAGGGCGGACTGAAGCTTTCTTT | GGACAAGGCACCAGACTCACAGTTGTAG | MGSRLLCWVLLCLLGA_ | _PS_ | KAGVTQTPRYLIKTRGQQVTLSCSPI | SGHRS | VSWYQQTPGQGLQFLFE | YFSETQ | RNKGNFPGRFSGRQFSNSRSEMNVSTLELGDSALYL | CASSREQTGRTEAFF | GQGTRLTVV_ | TATGCCCCTCCCACCAGGAAAAGCAAGGCTGAGAATTTAGCTCTTTCCCAGGAGGACCAAGCCCTGAGCACAGACACAGTGCTGCCTGCCCCTTTGTGCCATGGGCTCCAGGCTGCTCTGTTGGGTGCTGCTTTGTCTCCTGGGAGCAGGCCCAGTAAAGGCTGGAGTCACTCAAACTCCAAGATATCTGATCAAAACGAGAGGACAGCAAGTGACACTGAGCTGCTCCCCTATCTCTGGGCATAGGAGTGTATCCTGGTACCAACAGACCCCAGGACAGGGCCTTCAGTTCCTCTTTGAATACTTCAGTGAGACACAGAGAAACAAAGGAAACTTCCCTGGTCGATTCTCAGGGCGCCAGTTCTCTAACTCTCGCTCTGAGATGAATGTGAGCACCTTGGAGCTGGGGGACTCGGCCCTTTATCTTTGCGCCAGCAGCCGAGAGCAGACAGGGCGGACTGAAGCTTTCTTTGGACAAGGCACCAGACTCACAGTTGTAG | MGSRLLCWVLLCLLGAGPVKAGVTQTPRYLIKTRGQQVTLSCSPISGHRSVSWYQQTPGQGLQFLFEYFSETQRNKGNFPGRFSGRQFSNSRSEMNVSTLELGDSALYLCASSREQTGRTEAFFGQGTRLTVV_ |
| 5 | 5486 | 0.010589 | TRBV5-1 | TRBD1 | TRBJ2-7 | TRBC2 | TATGCCCCTCCCACCAGGAAAAGCAAGGCTGAGAATTTAGCTCTTTCCCAGGAGGACCAAGCCCTGAGCACAGACACAGTGCTGCCTGCCCCTTTGTGCC | ATGGGCTCCAGGCTGCTCTGTTGGGTGCTGCTTTGTCTCCTGGGAGCAG | GCCCAGTA | AAGGCTGGAGTCACTCAAACTCCAAGATATCTGATCAAAACGAGAGGACAGCAAGTGACACTGAGCTGCTCCCCTATC | TCTGGGCATAGGAGT | GTATCCTGGTACCAACAGACCCCAGGACAGGGCCTTCAGTTCCTCCTGGGG | TACTTCAGTGAGACACAG | AGAAACAAAGGAAACTTCCCTGGTCGATTCTCAGGGCGCCAGTTCTCTAACTCTCGCTCTGAGATGAATGTGAGCACCTTGGAGCTGGGGGACTCGGCCCTTTATCTT | TGCGCCAGCAGCTTGGAACAGGCAGCGCGCTCCTACGAGCAGTACTTC | GGGCCGGGCACCAGGCTCACGGTCACAG | MGSRLLCWVLLCLLGA_ | _PS_ | KAGVTQTPRYLIKTRGQQVTLSCSPI | SGHRS | VSWYQQTPGQGLQFLLG | YFSETQ | RNKGNFPGRFSGRQFSNSRSEMNVSTLELGDSALYL | CASSLEQAARSYEQYF | GPGTRLTVT_ | TATGCCCCTCCCACCAGGAAAAGCAAGGCTGAGAATTTAGCTCTTTCCCAGGAGGACCAAGCCCTGAGCACAGACACAGTGCTGCCTGCCCCTTTGTGCCATGGGCTCCAGGCTGCTCTGTTGGGTGCTGCTTTGTCTCCTGGGAGCAGGCCCAGTAAAGGCTGGAGTCACTCAAACTCCAAGATATCTGATCAAAACGAGAGGACAGCAAGTGACACTGAGCTGCTCCCCTATCTCTGGGCATAGGAGTGTATCCTGGTACCAACAGACCCCAGGACAGGGCCTTCAGTTCCTCCTGGGGTACTTCAGTGAGACACAGAGAAACAAAGGAAACTTCCCTGGTCGATTCTCAGGGCGCCAGTTCTCTAACTCTCGCTCTGAGATGAATGTGAGCACCTTGGAGCTGGGGGACTCGGCCCTTTATCTTTGCGCCAGCAGCTTGGAACAGGCAGCGCGCTCCTACGAGCAGTACTTCGGGCCGGGCACCAGGCTCACGGTCACAG | MGSRLLCWVLLCLLGAGPVKAGVTQTPRYLIKTRGQQVTLSCSPISGHRSVSWYQQTPGQGLQFLLGYFSETQRNKGNFPGRFSGRQFSNSRSEMNVSTLELGDSALYLCASSLEQAARSYEQYFGPGTRLTVT_ |
| 6 | 5438 | 0.010496 | TRBV5-1 | TRBD2 | TRBJ2-3 | TRBC2 | TATGCCCCTCCCACCAGGAAAAGCAAGGCTGAGAATTTAGCTCTTTCCCAGGAGGACCAAGCCCTGAGCACAGACACAGTGCTGCCTGCCCCTTTGTGCC | ATGGGCTCCAGGCTGCTCTGTTGGGTGCTGCTTTGTCTCCTGGGAGCAG | GCCCAGTA | AAGGCTGGAGTCACTCAAACTCCAAGATATCTGATCAAAACGAGAGGACAGCAAGTGACACTGAGCTGCTCCCCTATC | TCTGGGCATAGGAGT | GTATCCTGGTACCAACAGACCCCAGGACAGGGCCTTCAGTTCCTCTTTGAA | TACTTCAGTGAGACACAG | AGAAACAAAGGAAACTTCCCTGGTCGATTCTCAGGGCGCCAGTTCTCTAACTCTCGCTCTGAGATGAATGTGAGCACCTTGCTGCTGGGGGACTCGGCCCTTTATCTT | TGCGCCAGCAGCCTGGCTTTAGCGGGGACTAGCACAGATACGCAGTATTTT | GGCCCAGGCACCCGGCTGACAGTGCTCG | MGSRLLCWVLLCLLGA_ | _PS_ | KAGVTQTPRYLIKTRGQQVTLSCSPI | SGHRS | VSWYQQTPGQGLQFLFE | YFSETQ | RNKGNFPGRFSGRQFSNSRSEMNVSTLLLGDSALYL | CASSLALAGTSTDTQYF | GPGTRLTVL_ | TATGCCCCTCCCACCAGGAAAAGCAAGGCTGAGAATTTAGCTCTTTCCCAGGAGGACCAAGCCCTGAGCACAGACACAGTGCTGCCTGCCCCTTTGTGCCATGGGCTCCAGGCTGCTCTGTTGGGTGCTGCTTTGTCTCCTGGGAGCAGGCCCAGTAAAGGCTGGAGTCACTCAAACTCCAAGATATCTGATCAAAACGAGAGGACAGCAAGTGACACTGAGCTGCTCCCCTATCTCTGGGCATAGGAGTGTATCCTGGTACCAACAGACCCCAGGACAGGGCCTTCAGTTCCTCTTTGAATACTTCAGTGAGACACAGAGAAACAAAGGAAACTTCCCTGGTCGATTCTCAGGGCGCCAGTTCTCTAACTCTCGCTCTGAGATGAATGTGAGCACCTTGCTGCTGGGGGACTCGGCCCTTTATCTTTGCGCCAGCAGCCTGGCTTTAGCGGGGACTAGCACAGATACGCAGTATTTTGGCCCAGGCACCCGGCTGACAGTGCTCG | MGSRLLCWVLLCLLGAGPVKAGVTQTPRYLIKTRGQQVTLSCSPISGHRSVSWYQQTPGQGLQFLFEYFSETQRNKGNFPGRFSGRQFSNSRSEMNVSTLLLGDSALYLCASSLALAGTSTDTQYFGPGTRLTVL_ |
表头解释:
(1)Clone ID: 每种克隆编号,从1开始按顺序编号
(2)Clone Count: 每种克隆的总数
(3)Clone Fraction: 表示每种克隆占总TCR克隆种类的频率的百分比
(4)V/D/J/C Segment: VDJC基因片段ID
(5)V5UTR/L1/L2/FR1/CDR1/FR2/CDR2/FR3/CDR3/FR4 Gene Sequence: 全长克隆序列V5UTR/L1/L2/FR1/CDR1/FR2/CDR2/FR3/CDR3/FR4不同位置核酸序列
(6)L1/L2/FR1/CDR1/FR2/CDR2/FR3/CDR3/FR4 AA Sequence: 全长克隆序列L1/L2/FR1/CDR1/FR2/CDR2/FR3/CDR3/FR4不同位置氨基酸序列。
(7)FullSeq.Gene.Sequence: VDJ全长核酸序列
(8)FullSeq.AA.Sequence: VDJ全长氨基酸序列。
其中AA序列中出现的“*”和“_”,分别表示该区域片段有stop codon和out-of-frame
四、参考文献
[1].Wang, X., Hu, Y., Liu, X. et al. Quantitative characterization of T-cell repertoire alteration in Chinese patients with B-cell acute lymphocyte leukemia after CAR-T therapy. Bone Marrow Transplant 54, 2072–2080 (2019).
[2].Xiaoyue Gao, Chen Xu, Botao Li, et al. Quantitative Analysis of Thymus-independent Donor-derived T Cell Expansion in Transplant Patients. Biology of Blood and Marrow Transplantation. VOLUME 26, ISSUE 2, P242-253, FEBRUARY 01, 2020.
[3].Xiao Liu, Girish Venkataraman, Jiaying Lin , et al. Highly clonal regulatory T-cell population in follicular lymphoma – inverse correlation with the diversity of CD8+ T cells, OncoImmunology, 4:5.
[4].John F. Beausang, Amanda J. Wheeler, Natalie H. Chan, et al. T cell receptor sequencing of early-stage breast cancer tumors identifies altered clonal structure of the T cell repertoire. PNAS Nov 2017, 114 (48) E10409-E10417;.
[5].Tumeh PC, et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance. Nature.2014;515:568–571. doi: 10.1038/nature13954.
[6].Johnson D B, Frampton G M, Rioth M J, et al. Targeted next generation sequencing identifies markers of response to PD-1 blockade[J]. Cancer immunology research, 2016, 4(11): 959-967.
[7].Reuben A, Gittelman R, Gao J, et al. TCR repertoire intratumor heterogeneity in localized lung adenocarcinomas: an association with predicted neoantigen heterogeneity and postsurgical recurrence[J]. Cancer discovery, 2017, 7(10): 1088-1097.
[8].A note on a generalization of Pielou's equation.
[9].On a generalization of the global attractivity for a periodically forced Pielou's equation.
[10].On the relationship between Pielou's evenness and landscape domi v nance within the context of Hill's diversity profiles.
[11].Shannon, C. E. The mathematical theory of communication. 1963.MD Comput. 14, 306–317 (1997).
[12].Ruggiero E, Nicolay J P, Fronza R, et al. High-resolution analysis of the human T-cell receptor repertoire[J]. Nature communications, 2015, 6: 8081.
[13].Jia Q, Wu W, Wang Y, et al. Local mutational diversity drives intratumoral immune heterogeneity in non-small cell lung cancer[J]. Nature Communications, 2018, 9.
[14].Simpson, E. H. Measurement of diversity. Nature 163, 688 (1949).
[15].Jia Q, Wu W, Wang Y, et al. Local mutational diversity drives intratumoral immune heterogeneity in non-small cell lung cancer[J]. Nature Communications, 2018, 9.
[16].Van Heijst J W J, Ceberio I, Lipuma L B, et al. Quantitative assessment of T cell repertoire recovery after hematopoietic stem cell transplantation[J]. Nature medicine, 2013, 19(3): 372.
[17].Ruggiero E, Nicolay J P, Fronza R, et al. High-resolution analysis of the human T-cell receptor repertoire[J]. Nature communications, 2015, 6: 8081.
[18].Maximum Likelihood Estimation of Functionals of Discrete Distributions.2014.
[19].Information, Measurement, and Quantum Mechanics.1987.
[20].Postow M A, Manuel M, Wong P, et al. Peripheral T cell receptor diversity is associated with clinical outcomes following ipilimumab treatment in metastatic melanoma[J]. Journal for immunotherapy of cancer, 2015, 3(1): 23.
[21].Hogan S A, Courtier A, Cheng P F, et al. Peripheral blood TCR repertoire profiling may facilitate patient stratification for immunotherapy against melanoma[J]. Cancer immunology research, 2019, 7(1): 77-85.
[22].Briney,B., Inderbitzin,A., Joyce, C., & Burton, D. R. (2019). Commonality despite exceptionaldiversity in the baseline human antibody repertoire. Nature, 566(7744),393–397.
[23].Liang Q, Liu Z, Zhu C, Wang B, LiuX, Yang Y, LvX, Mu H, Wang K. Intrahepatic T-Cell Receptor β ImmuneRepertoire Is Essential for Liver Regeneration.Hepatology.2018 Nov;68(5):1977-1990.
[24].Miho,E., Yermanos,A., Weber, C. R., Berger, C. T., Glanville, J., & Greiff,V. (2018). Computational Strategies for Dissecting the High-DimensionalComplexity of Adaptive immune Repertoires. Frontiers in Immunology,9(February), 1–15.
[25].Sheng J, Wang H, Liu X, Deng Y, Yu Y, Xu P, Shou J, Pan H, Li H, Zhou X, Han W, Sun T, Pan H, Fang Y. Deep Sequencing of T-Cell Receptors for Monitoring Peripheral CD8+ T Cells in Chinese Advanced Non-Small-Cell Lung Cancer Patients Treated With the Anti-PD-L1 Antibody. Front Mol Biosci. 2021 Jul 9;8:679130.
[26].Briney, B., Inderbitzin, A., Joyce, C. et al. Commonality despite exceptional diversity in the baseline human antibody repertoire. Nature 566, 393–397 (2019).
[28].Wang, X. et al. Quantitative characterization of T-cell repertoire alteration in Chinese patients with B-cell acute lymphocyte leukemia after CAR-T therapy. Bone marrow transplantation, doi:10.1038/s41409-019-0625-y (2019).
[29].Gao, X. et al. Quantitative Analysis of Thymus-Independent Donor-Derived T Cell Expansion in Transplant Patients. Biology of blood and marrow transplantation : journal of the American Society for Blood and Marrow Transplantation 26, 242-253, doi:10.1016/j.bbmt.2019.10.026 (2020).
[30].Mingyao Pan 1, T cell receptor convergence is an indicator of antigen-specific T cell response in cancer immunotherapies. Elife. 2022 Nov 9:11:e81952.
[31].Timothy John Looney, TCR Convergence in Individuals Treated With Immune Checkpoint Inhibition for Cancer. Front Immunol. 2019; 10: 2985.
[32].Yang, S., Xu, J., Dai, Y. et al. Neutrophil activation and clonal CAR-T re-expansion underpinning cytokine release syndrome during ciltacabtagene autoleucel therapy in multiple myeloma. Nat Commun 15, 360 (2024).