
发表期刊:Frontiers in Immunology 发表时间:2023年8月4日 影响因子:7.3 研究方法:收集11例乳腺癌患者的活检组织样本,进行体外培养,大约10天后,采集肿瘤浸润淋巴细胞(称为TILs-initial),通过流式细胞术评估CD4+ 和CD8+ TILs 的丰度,同时收集一部分细胞悬液进行TCR测序和分析;在最初培养后的第2至4周,从一些TILs中分选和扩增CD4+、CD8+T细胞,称为TILs-CD4+ 和TILs-CD8+,进行TCR分析(图1)。 采集6名健康志愿者的外周血PBMC作为对照,一部分使用bulk细胞以及分选的CD4+和CD8+细胞(分别称为PBMC,P-CD4+和P-CD8+)直接用于TCR分析;其余部分采用和TILs同样的方法培养和扩增,分别称为ePBMC、eP-CD4+ 和eP-CD8+,进行TCR测序分析。
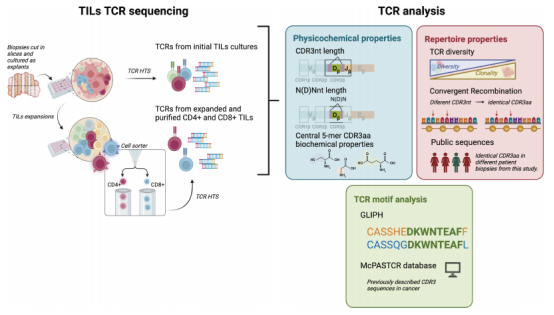
图1.CD4+和CD8+TILs TCR库分析 作者分析了CD4+和CD8+ TILs中CDR3核酸(nt)和N(D)Nnt区域的序列长度以及CDR3氨基酸(aa)序列的生化性质。TILs-initial样本和TILs-CD4+、TILs-CD8+样本的TRB CDR3nt长度差异不显著(图2A)。但是TILs-CD8+样本中TRB N(D)Nnt 的平均长度 (11.4 ± 2.2) 显著短于TILs-CD4+样本(14.4 ± 3.1, p = 0.004) 和TILs-initial样本(13.8 ± 2.1, p = 0.019)(图2B)。在对照样本中,无论是P-CD4+和P-CD8+样本还是eP-CD4+和eP-CD8+样本,都没有观察到TRB N(D)Nnt序列长度的显著差异(图2C、D)。 然后作者分析了β链CDR3中心5个氨基酸的生化性质(亲水性指数、电荷和极性)(图2E、F)。在这三种特性中,只观察到TRB序列的电荷差异,在TILs-CD4+样本中平均值最高,与TILs-CD8+样本和TILs-initial样本有显著差异(图2E)。这表明,在TILs-CD4+中,β链CDR3中心5-mer中正电荷氨基酸的存在增加。在对照分析中发现P-CD4+和P-CD8+的电荷存在显著差异(图2F)。 因此,作者得出结论,TRB N(D)Nnt长度的差异是TILs的一个特征,而健康人群的外周T细胞没有该差异,这可能表明不同TIL亚群中CDR3长度的结构变化与不同pMHC复合物的亲和力以及它们在肿瘤微环境中的可用性有关。而CDR3aa中心5-mer中使用正或负带电氨基酸的差异是CD4+和CD8+细胞固有的TCR特征。
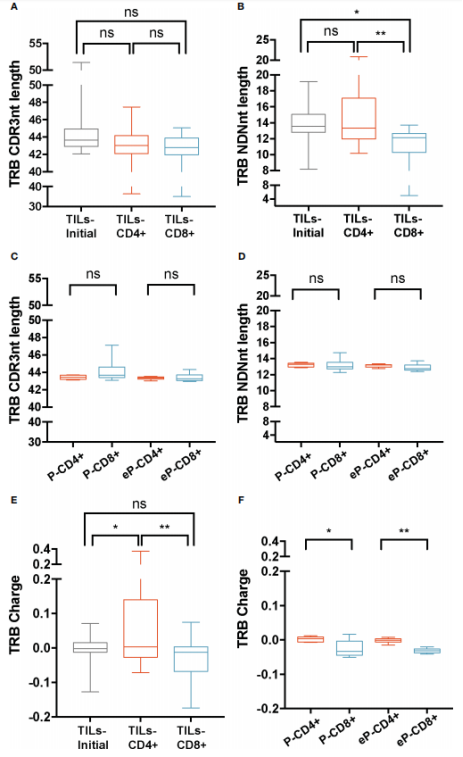
图2.来自TILs和健康人外周T细胞的TRB理化特性 不同T细胞亚群之间的TCR多样性可能有所不同,反映了CD4+和CD8+T细胞之间抗原识别能力的差异。因此,作者研究了TILs-CD4+和TILs-CD8+样本中的TCR多样性,使用归一化Shannon-Wiener指数(nS-W)。 TILs-initial样本与TILs-CD4+、TILs-CD8+样本的TRB CDR3nt序列的多样性没有显著差异(图3A)。这可能是TCR多样性值的广泛分散掩盖了真实的多样性差异,因此作者根据活检切片对数据进行了分层,然后比较了两个子集中的多样性指数。通过这种方法,TILs-CD8+样本显示出比TILs-CD4+样本更高的多样性(图3B)。 作者使用初始培养物中两组亚群的比例来推断不同程度的多样性与亚群比例之间是否存在相关性。发现多样性与CD4比例呈负相关且显著相关(与CD8呈正相关但不显著),表明在初始培养物扩增之前已经存在多样性差异(图3D、E)。因此,作者认为,CD4+ TILs具有较低多样性的TCR库,当CD4+ T细胞比例较高时,这一特征可以在初始培养物中观察到。 同时,分析对照样本中的TCR多样性(图3C).在PBMC和e-PBMCs样本中均未发现显著差异。
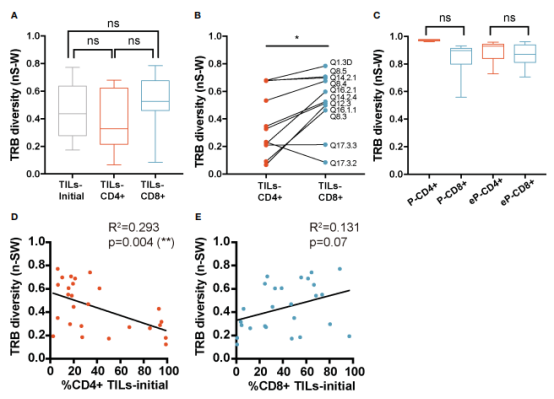
图3.TILs和健康供体外周T细胞中的TRB多样性 3、TILs-CD4+中发现的较低多样性不能归因于公共序列的扩增或较高的趋同性 较低的多样性或较高的克隆性可能是由于在肿瘤浸润中选择某些特定克隆型所致。因此,在TILs-CD4+样本中观察到的较低多样性可能表明该亚群中的选择更受限,这可能是更高程度的趋同重组(convergent recombination,CR)的结果,即由于免疫反应期间的TCR选择,导致多个CDR3nt编码相同的CDR3aa序列。 在活检样本中,与TILs-initial样本相比,TILs-CD4+和TILs-CD8+的TRB序列趋同(convergence)性显著增加(图4A),这表明体外扩增可能通过在不同的活检切片培养物中选择某些克隆型来影响趋同水平。在对照分析中,没有发现不同样品之间的CR水平有任何差异(图4B). 由于CR水平不能解释TILs-CD4+样本中观察到的较低多样性,作者研究了是否是由于公共序列比例较高所致(图4C)。将至少在两个活检/患者中共享的称为公共(public)序列,而仅在一个活检中发现的则称为私有(private)序列。TILs-CD4+和TILs-CD8+样本中的公共序列分别有17个和12个,仅占总TRB CDR3aa序列的0.4%。 作者还发现大部分私有序列是非趋同性的,即由唯一的CDR3nt序列编码,而大部分公共序列则是趋同性的(图4 E)。因此,作者认为,CR水平与公共序列的比例有关,但这些特征都与TCR多样性无关。 若想了解更多关于convergence的解读,可参考文章TCR测序新指标:免疫治疗实战利器,从疗效到预后全面解析
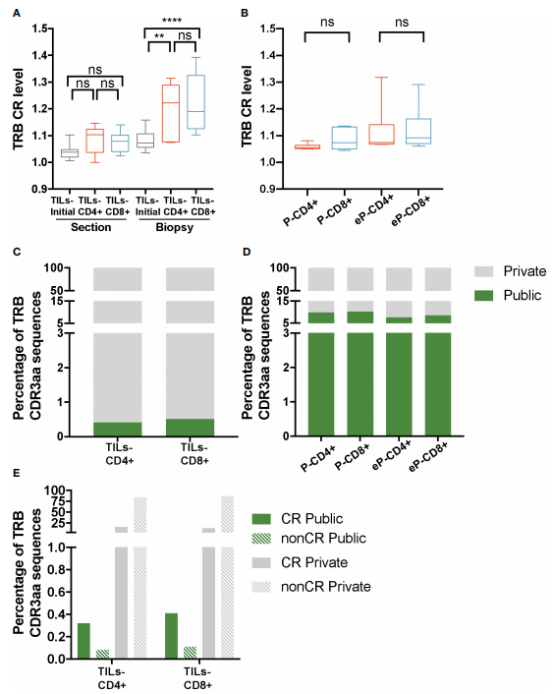
图4.TILs和健康人外周T细胞中的TRB CR水平及公共序列占比 4、扩增的TILs-CD4+呈现相似的TCR,在患者中共享保守的CDR3aa motifs TCR相似性可能是由于CDR3aa序列中常见的3-5-mer基序(motif)。根据既往文献中提到的GLIPH2算法通过在 CDR3上聚类短氨基酸基序来识别具有高概率共享抗原特异性的TRB CDR3aa序列簇。作者应用GLIPH2,在TILs-CD4+中鉴定出的基序数量明显大于TILs-CD8+(45 对25)(图5A).几个基序在相当一部分TCR中存在,并且在两种样本亚型中按样本量归一化后分布相似(图 5B,C).
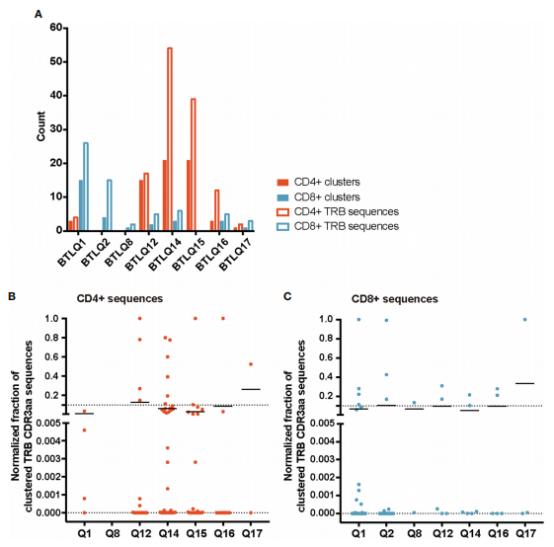
图5.TILs-CD4+和TILs-CD8样本中的TRB CDR3aa基序 在TILs-CD4+样本检测到的45个基序中,有15个存在于至少两种不同的活检中(图6A).一些基序出现在五个CDR3aa序列中,甚至超过十个(图6B)。在TILs-CD8+样本中,只有4个基序在一个以上的活检样本中发现(图6C),最常见的基序出现在7个CDR3aa序列中(图6D).因此作者得出结论,CD4+ TILs不仅多样性较低,而且其CDR3aa序列在个体内部和个体间也更加相似。TILs-CD8+样本中基序数量较多的两个活检样本对应于TN型病例。
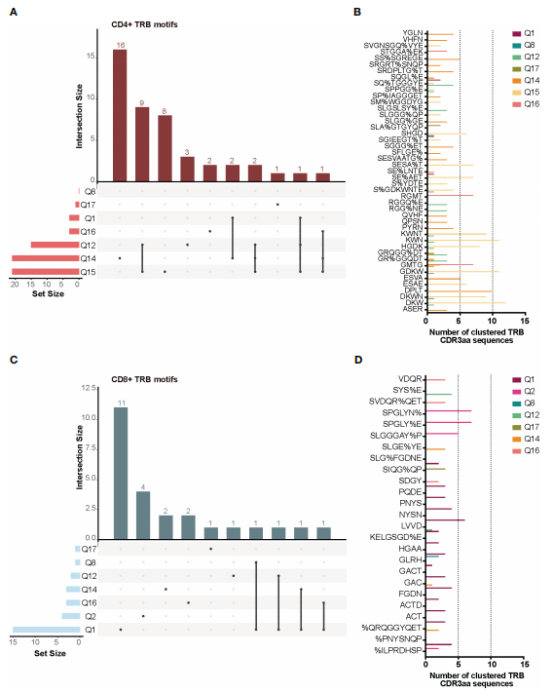
图6.TRB CDR3aa共享基序和氨基酸序列 5、TCR 基序可用于鉴定肿瘤反应性CD4+ T细胞 作者推断在TILs-CD4+样本中观察到的个体内及个体间的相对同质性可能与癌症相关的扩增有关。将该研究的TCR CDR3aa数据的三类序列,即频率>0.1%的序列、公共序列和由GLIPH基序聚类的序列,与McPAS-TCR(一个已知特异性/病理背景的TCR序列的综合数据库)中报告的序列进行了比较。 在TILs-CD4+中,基序聚类的序列在McPAS-TCR中的匹配数量大于高频或公共序列的匹配数量,但在TILs-CD8+中,频率>0.1%序列获得的匹配数量更大。通过手动注释,TILs-CD4+和TILs-CD8+样本中与癌症基序相关的序列数分别增加到10 个和13 个(表1),它们以前都没有在乳腺癌中报道过(图7A、B)。 在TILs-CD4+中,四个基序中有两个与在两种不同活检中发现的序列相关,但在TILs-CD8+组中,每个序列簇仅与一种活检相关(图7C,D)。 在TILs-CD4+样本中,基序聚类比使用频率> 0.1%和公共序列聚类提供了更多的匹配。因此,基序分析作为一种选择方法具有优势,可以增加候选序列的数量,并且可能特别适用于鉴定肿瘤反应性CD4+克隆型。
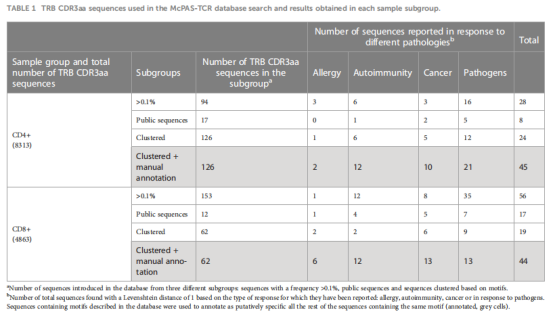
表1.和McPAS-TCR数据库的匹配结果
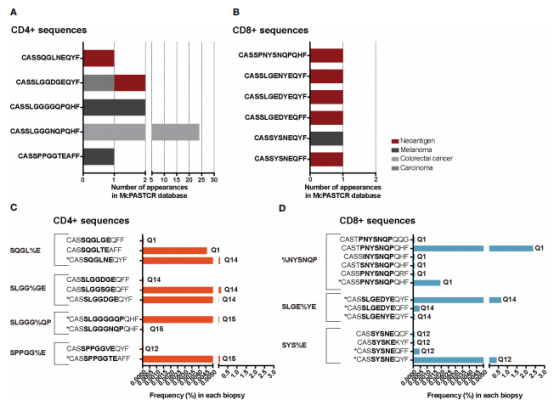
图7.在McPAS-TCR数据库中发现的TRB CDR3序列和使用基序注释的序列 该研究证明CD4+和CD8+ T细胞的TCR库之间存在差异,这可能与它们的识别能力和细胞功能有关。作者发现CD4+ T细胞反应在个体之间更加均匀,而CD8+ T细胞反应更局限于免疫原性更强的肿瘤,如TNBC。这在选择基于驱动免疫反应的抗原的有效个性化治疗时可能特别重要:I 类和 II 类限制性 TCR 可分别用于靶向肿瘤特异性和肿瘤相关抗原。通过对TILs中TCR序列的全面剖析,有望推动更精准的免疫细胞疗法研发,以期提高临床疗效并优化患者治疗方案。 参考文献 Aran A, Lázaro G, Marco V, Molina E, Abancó F, Peg V, Gión M, Garrigós L, Pérez-García J, Cortés J, Martí M. Analysis of tumor infiltrating CD4+ and CD8+ CDR3 sequences reveals shared features putatively associated to the anti-tumor immune response. Front Immunol. 2023 Aug 4;14:1227766. doi: 10.3389/fimmu.2023.1227766. 杭州艾沐蒽生物科技有限公司成立于2016年,是国际前沿的专注于免疫驱动医学技术的国家高新技术企业。创始人团队来自美国芝加哥大学,在2010年开始使用免疫组基因高通量测序技术开展各种疾病相关研究,于2016年通过自主研发,全国首家推出NGS-MRD血液肿瘤微小残留病(MRD)检测Seq-MRD®,并授权泛生子使用。同时,公司拥有Immun-Traq®肿瘤治疗伴随诊断、Immun-Cheq® |T细胞免疫测评以及ImmuHub®免疫组测序科研服务产品,并布局有基于AI机器学习算法的T-classifier®肿瘤早筛、单细胞测序、TCR-T和抗体发现等平台管线。目前为止发表了数十篇论文,其中包括:The New England Journal of Medicine(IF:158.5), Nature(IF:65), Signal Transduction and Targeted Therapy(IF:40), Cellular and Molecular Immunology(IF:24), Nature Communications (IF:17)等多篇高分杂志。公司构建几十项发明专利和软件著作权为核心的自主知识产权体系,为医院临床、生命科学研究、新药开发等提供解决方案和产品。艾沐蒽专注于通过解码适应性免疫系统来改变疾病的诊断和治疗,并致力于推进免疫驱动医学领域发展。
