肿瘤早筛早诊是世界性难题,尤其是发病率、死亡率高的恶性肿瘤,早期诊断和治疗能极大改善患者的生存结局。艾沐蒽T-Classifier®平台致力于将T细胞受体(TCR)二代测序(NGS)与人工智能(AI)机器学习相结合,实现多种恶性肿瘤的早筛早诊,其原理是利用TCR能极其敏感且特异性识别肿瘤抗原的特性,通过机器学习算法找到肿瘤特异性TCR的相关特征,建立诊断模型。 2024年7月4日,艾沐蒽联合内蒙古大学数学科学学院达林教授团队在Nature子刊Scientific Reports杂志发表题为“Comprehensive application of AI algorithms with TCR NGS data for Glioma diagnosis”的文章,该研究利用艾沐蒽T-Classifier®平台为胶质瘤(Glioma)的诊断提供了一种新的高准确率方法,这是AI技术在癌症诊断中的一次成功实践,为其他癌症的诊断和治疗提供了新的思路。

首先从两个不同的实验中获得两组胶质瘤样本TCR测序数据,并选择相应数量的健康供体(HD)样本形成本实验的训练和测试数据。对测序数据进行Fisher精确检验,得到不同序列中胶质瘤患病率对应的P值。根据P值不同指数水平阈值的选择,得到相应的相关序列集,通过这些序列集,得到三种不同类型的数据结构,并引入分类算法,分为4个实验,如下图所示。

分析了所有样本的6个克隆性和多样性指数,作者采用了GBDT(梯度提升决策树)、RF(随机森林)、XGBoost(极端梯度提升)、排列等四种特征选择方法来对六个指标的重要性进行排序,相应的条形图如图1A至图1D所示。图1E和图1F分别表示了从四种特征选择方法得出的重要性之和以及重要性等级之和。很明显,VJ多样性指数(Hvj.Index)的重要性最高,其次是单克隆T细胞比例(Singleton)和Simpson多样性指数(Invsimpson.Index)。

图1 图1G展示了各指标与表型的相关性。重要性较高的三个指标与表型的相关性更强;相反,重要性最低的指标克隆性指数(Clonality index)与表型的相关性最弱。利用这三个相关的多样性指标,我们可以获得良好的分类AUC,如图1H中的ROC曲线所示,Random Forest、XGBoost 和 Bagging 分类器都实现了100%的分类AUC值。图1I-图1K显示出胶质瘤患者与健康人各指数的明显差异。

图1 虽然基于多样性指标的分类算法能够获得良好的分类AUC,但是既往有研究发现TCR多样性在其他癌种(比如肺癌)和健康人中也具有明显差异,说明基于多样性指数的分类方法癌种特异性有限,因此作者计划利用TCR序列分布和存在数据的准确使用,找到胶质瘤特异性TCR特征来进行分类。 利用二维数据特征(总克隆类型和相关克隆类型),作者构建了包含神经网络算法、集成学习算法和传统机器学习算法在内的16种算法的分类系统。图2A说明二维特征尤其是相关克隆型的数量与表型显着相关。与HD相比,胶质瘤患者拥有更多的相关克隆型(图2C)。基于这些发现,作者采用多种算法基于二维特征构建分类器,所有算法均实现了优异的分类性能,一些算法甚至实现了AUC为1,这凸显了二维特征提取方法诊断胶质瘤的有效性(图2D)。在图2E-J 中,给出了与几种表现出良好分类结果的算法相对应的决策边界。


图2 使用了多种分类算法分析多维数据集,包括随机森林、具有线性和多项式核函数的支持向量机、逻辑回归、线性判别分析、贝叶斯算法、GBDT、XGB和决策树算法。通过对Fisher精确检验应用不同的临界值,确定了与神经胶质瘤具有不同程度相关性的序列组。然后将这些序列合并到分类算法系统中,以使用更少的序列数量实现更高的分类精度。 图3A显示了健康和神经胶质瘤患者组之间克隆种类的交集,只有一小部分是两者共有的。众多算法中线性判别分析的准确度最高,AUC为100%(图3B)。如图3C所示,每列的二进制数据(0或1),指示相应样品中是否存在特定的TCRβ序列,通过分析序列,确定了一组与神经胶质瘤直接相关的TCRβ序列,并利用少量序列准确预测疾病状态。 然后将这些序列数据集应用于12种分类算法,通过选择合适的P值,将疾病相关序列缩小到12个,图3D显示表型与这些序列具有相对较高的相关性。通过仅利用这12个 TCRβ序列,实现了高预测准确率。图3E展示了P值为0.001时这12个序列的重要性排序。
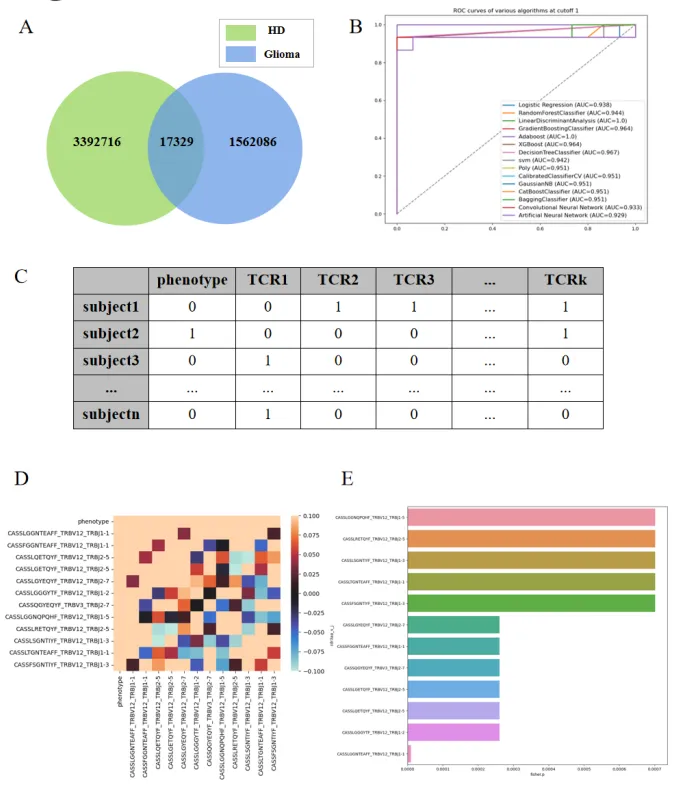
图3 除了Fisher的精确检验外,本研究还利用Lasso和RFECV两种算法进行特征提取。作者采用Lasso算法从通过 Fisher精确检验获得的多维数据中选择特征,阈值为 0.1、0.01 和 0.001。本研究将RFECV与9种分类算法相结合进行特征提取,分别使用12种分类算法评估特征提取的有效性。这种方法改进了特征提取,以更少的序列产生了更高的分类精度。 图4A和图4B显示了Lasso的重要性分布。阈值0.1产生了 19个序列特征,而阈值0.01和0.001分别产生了11个和9个序列特征。相对而言,序列仍然太多。而在RFECV算法方面,如图4C所示,采用的特征提取算法为Adaboost(自适应提升),使用的分类算法为SVM(支持向量机)。这种方法产生的特征数为5,AUC和精度均为 0.967。在这 5 个序列中,使用排除剔除法进一步提取后,剩余三个特征序列,分别为 CASSLGGNTEAFF_TRBV12_TRBJ1-1、CASSYSDTGELFF_TRBV6_TRBJ2-2 和 CASSLTGNTEAFF_TRBV12_TRBJ1-1。通过这三个核心序列,可以获得高AUC和低相关度(图4E、4G)。 使用相同的方法分析了阈值为0.001的数据集,该数据集由 12个序列的组合组成。如图4D所示,采用的特征提取算法为Adaboost,使用的分类算法为CV(CalibratedClassifierCV),该分析产生了五个序列。通过这五个序列,AUC为0.967,准确度为0.933,特异性为1。同样,通过逐个筛选,确定了三个序列的组合:CASSLGETQYF_TRBV12_TRBJ2-5、CASSLGGNQPQHF_TRBV12_TRBJ1-5和CASSLSGNTIYF_TRBV12_TRBJ1-3。同时,图4F和图4H表明,这三个相关性较低的序列的组合可以对AUC值进行分类并获得高分类AUC值。


图4 本研究通过分析来自神经胶质瘤患者和健康个体的高通量测序数据,探索TCR序列多样性指数的多维分类和特征选择,以及TCR相关序列的二维分类和特征选择分析。从这些分析中确定了两组核心序列,足以在胶质瘤检测中达到 96.7% 的诊断准确率。 我们的研究证明了T-Classifier®不仅适用于神经胶质瘤诊断,而且适用于诊断其他病毒(如CMV,详见艾沐蒽联合发表Nature子刊|T-Classifier®AI诊断模型准确率92.86%)以及各种类型的癌症。这种方法对未来的癌症早筛早诊有很大的希望,艾沐蒽未来的工作将侧重于扩大该方法的应用,实现多个癌种的早期筛查和诊断。 文献来源:Zhou, K., Xiao, Z., Liu, Q. et al. Comprehensive application of AI algorithms with TCR NGS data for glioma diagnosis. Sci Rep 14, 15361 (2024).
